ТРАНСКРИБИРОВАТЬ — это… Что такое ТРАНСКРИБИРОВАТЬ?
- ТРАНСКРИБИРОВАТЬ
- ТРАНСКРИБИРОВАТЬ
- ТРАНСКРИБИ́РОВАТЬ, транскрибирую, транскрибируешь, совер. и несовер., что (спец.). Произвести (производить) транскрипцию чего-нибудь. Транскрибировать иностранную фамилию русскими буквами.
Толковый словарь Ушакова. Д.Н. Ушаков. 1935-1940.
.
- ТРАНСКРИБИРОВАННЫЙ
- ТРАНСКРИБИРОВАТЬСЯ
Смотреть что такое «ТРАНСКРИБИРОВАТЬ» в других словарях:
транскрибировать — рую, рует, несов., что (нем. transkribieren … Словарь иностранных слов русского языка
Транскрибировать — I несов.

транскрибировать — транскриб ировать, рую, рует … Русский орфографический словарь
транскрибировать — (I), транскриби/рую, руешь, руют … Орфографический словарь русского языка
транскрибировать — рую, руешь; св. и нсв. (св. также протранскрибировать). что. Спец. Произвести производить транскрипцию. Т. текст. Т. немецкое название русскими буквами. Т. фортепьянную пьесу для скрипки. ◁ Транскрибироваться, руется; страд. Транскрибирование, я; … Энциклопедический словарь
транскрибировать — рую, руешь; св. и нсв. (св., также, протранскриби/ровать) см. тж. транскрибироваться, транскрибирование что спец. Произвести производить транскрипцию.
Транскриби/ровать текст. Тран … Словарь многих выражений
транскрибировать(ся) — транскриб/ир/ова/ть(ся) … Морфемно-орфографический словарь
Транскрибирование — I ср. 1. процесс действия по несов. гл. транскрибировать I 2. Результат такого действия. II ср. 1. процесс действия по несов. гл. транскрибировать II 2. Результат такого действия. Толковый словарь Ефремовой. Т. Ф. Ефремова. 2000 … Современный толковый словарь русского языка Ефремовой
Транскрибироваться — I несов. неперех. страд. к несов. гл. транскрибировать I II несов. неперех. страд. к несов. гл. транскрибировать II Толковый словарь Ефремовой. Т. Ф. Ефремова. 2000 … Современный толковый словарь русского языка Ефремовой
Азбука
 А. Брокгауза и И.А. Ефрона
А. Брокгауза и И.А. Ефрона
Транскрибирование бесед (предварительная версия) — служба «Речь» — Azure Cognitive Services
- Чтение занимает 3 мин
В этой статье
Транскрибирование бесед — это решение для преобразования речи в текст, объединяющее в себе распознавание речи, идентификацию говорящего и соотнесение предложений с каждым говорящим (также известное как диаризация) для транскрибирования любой беседы в реальном времени и (или) асинхронного транскрибирования. Транскрибирование бесед различает участников беседы, чтобы определить, кто, что и когда произнес, и упрощает для разработчиков добавление преобразования речи в текст в приложения, выполняющие диаризацию для нескольких говорящих.
Основные возможности
- Метки времени: речевой фрагмент каждого говорящего имеет метку времени, что позволяет без труда определять время произнесения фразы.
- Удобные для чтения расшифровки: в расшифровки автоматически добавляется форматирование и пунктуация, чтобы текст точно соответствовал беседе.
- Профили пользователей: профили пользователей создаются путем сбора образцов голосов пользователей и последующей их отправки для создания подписей.
- Диаризация для нескольких говорящих: позволяет определить, кто что сказал, путем синтеза звукового потока с помощью идентификатора каждого говорящего.
- Транскрибирование в реальном времени: предоставляет расшифровки, позволяющие понять, кто, что и когда произносит, во время беседы.

- Асинхронное транскрибирование: предоставляет более точные расшифровки благодаря использованию многоканального звукового потока.
Примечание
Несмотря на то что транскрибирование бесед не ограничивает число говорящих в комнате, оно оптимизировано для 2–10 говорящих за сеанс.
Начало работы
Чтобы приступить к работе, ознакомьтесь с кратким руководством по транскрибированию бесед в реальном времени.
Варианты использования
Чтобы принимать участие в собраниях могли все, в том числе, например, лица с нарушениями слуха, важно обеспечить транскрибирование в реальном времени. Транскрибирование бесед в режиме реального времени принимает аудиозапись собрания и определяет, кто что говорит, благодаря чему все участники собрания могут следить за расшифровкой и без задержки принимать участие в собрании.
Повышение эффективности
Участники собрания могут сосредоточиться на собрании, а ведение записей оставить решению транскрибирования бесед. Участники могут активно принимать участие в собрании и быстро переходить к дальнейшим действиям с помощью расшифровки. Самостоятельно вести во время собрания записи, в которых сложно ничего не упустить, больше не нужно.
Участники могут активно принимать участие в собрании и быстро переходить к дальнейшим действиям с помощью расшифровки. Самостоятельно вести во время собрания записи, в которых сложно ничего не упустить, больше не нужно.
Принцип работы
Это общий обзор работы решения для транскрибирования бесед.
Ожидаемые входные данные
Примечание
Образцы голосов пользователей не являются обязательными. Без этих входных данных в расшифровке будут отображаться разные говорящие, однако вместо имен конкретных говорящих, которые были зарегистрированы заранее и распознаны, будут использоваться наименования Speaker1, Speaker2 и т. д.
Транскрибирование в реальном времени и асинхронное транскрибирование
Транскрибирование бесед предлагает три режима расшифровки.
В режиме реального времени
Звуковые данные обрабатываются в реальном времени, чтобы вернуть идентификатор говорящего и расшифровку. Этот режим следует выбирать, если решение транскрибирования требует предоставления участникам беседы интерактивной расшифровки текущей беседы. Например, создание приложения с целью повышения удобства собраний для глухих и лиц с нарушениями слуха — это идеальный вариант использования транскрибирования в реальном времени.
Например, создание приложения с целью повышения удобства собраний для глухих и лиц с нарушениями слуха — это идеальный вариант использования транскрибирования в реальном времени.
Асинхронный
Выполняется пакетная обработка звуковых данных для возврата идентификатора говорящего и расшифровки. Этот режим следует выбирать, если решение транскрибирования требует обеспечить более высокую точность без представления расшифровки в реальном времени. Например, если вы хотите создать приложение, позволяющее участникам собрания без проблем получать информацию с пропущенных собраний, используйте режим асинхронного транскрибирования для получения результатов расшифровки высокой точности.
Транскрибирование в реальном времени и асинхронное транскрибирование
Звуковые данные обрабатываются в реальном времени для возврата идентификатора говорящего и расшифровки. Кроме того, создается запрос на получение расшифровки высокой точности путем асинхронной обработки. Этот режим следует выбирать, если приложению требуется расшифровка в реальном времени, а также расшифровка более высокой точности для использования после беседы или собрания.
Поддержка языков
В настоящее время транскрибирование бесед поддерживает все языки преобразования речи в текст в следующих регионах: eastasia, eastus, westeurope. Если требуется поддержка дополнительных языковых стандартов, обратитесь к специалистам по функции транскрибирования бесед.
Дальнейшие действия
4 правила дословной транскрибации | Бюро переводов СВАН
Четыре правила дословной транскрибации
Дословная транскрибация – это искусство преобразования устной речи в текст, отображающее речь в тексте точно так, как она звучала.
Для транскрибирования необходимы острый слух и внимание к деталям. Дословная транскрибация не может быть результатом механического слушания и печатания. Необходимо уделять внимание каждому звуку, интонации, слову и разумно пользоваться пунктуацией для надлежащей передачи смыслов.
Например:
Женщина: «Мы получили большую поддержку от…ммм… от коммунальных служб» (начинает плакать ребёнок).
Интервьюер: «Может быть, вы хотите ненадолго прерваться»?
Женщина: «Нет, всё в порядке, мне просто нужно его перевернуть (говорит с ребенком в течение 60 секунд). Так вот, я говорила…, ммм…мы получили большую поддержку в течение нескольких первых недель, после того как я вышла из больницы».
В этом примере транскрибация содержит слова, а также другие детали, например, фоновые шумы, упоминание о том, как мать говорит с ребёнком, и «холостые сообщения».
В этом и состоит смысл дословной транскрибации – необходимо записывать не только то, что было сказано, но также и как это было сказано и в каких обстоятельствах.
Типы дословной транскрибации.
В то время как истинным смыслом дословной транскрибации остается точная передача того, что происходит на записи, со временем выработались различные её виды, предназначенные для удовлетворения различных потребностей.
Различают в основном три вида дословной транскрибации:
- Интеллектуальная дословная транскрибация.

- Дословная транскрибация.
- Настоящая дословная транскрибация.
У каждого типа транскрибации имеются свои преимущества для удовлетворения различных потребностей. Давайте рассмотрим каждый тип подробнее.
1. Интеллектуальная дословная транскрибация
Интеллектуальная дословная транскрибация (известная также как хорошо читаемая дословная транскрибация) – это транскрибация с подробной редактурой и возможной перестановкой некоторых фраз, с целью сделать транскрибацию удобочитаемой.
Этот стиль идеально подходит для получения транскрибаций, не содержащих ошибок и готовых к печати. Интеллектуальная дословная транскрибация больше всего подходит для бизнес-целей.
Что содержит в себе транскрибация: всё, что сказано на записи, с небольшим парафразом (при необходимости).
Что не содержит в себе транскрибация: грамматических ошибок, «холостых сообщений» (междометий, «вы знаете» и пр.), запинаний, незавершённых предложений, повторов, невербальных сообщений и звуков окружающей среды.
2. Дословная транскрибация
Дословная транскрибация отличается от интеллектуальной дословной транскрибации чуть большей точностью.
При дословной транскрибации записывается каждое слово, присутствующее на записи, запись ведётся в режиме «как есть», включая грамматические ошибки и запинки. Однако, заминки и повторы при этом исключаются.
Этот тип транскрибации предпочтителен для студентов, исследователей и журналистов, которым необходимо, чтобы текст точно соответствовал записи речи, но при этом не включал в себя ненужные детали.
Что содержит в себе транскрибация: каждое слово, присутствующее на записи речи, в том числе заминки, уместные повторы и грамматические ошибки.
Что не содержит в себе транскрибация: «холостых сообщений», заминок, неуместных повторений, невербальной коммуникации и шумов окружающей среды.
3. Настоящая дословная транскрибация
Настоящая дословная транскрибация – это наиболее точная и подробная транскрибация записи речи, включающая в себя каждое слово и невербальную коммуникацию.
Поскольку эта транскрибация наиболее точная и подробная, то она предпочтительна для академических исследований и аналитических материалов, где значение имеет каждая мелочь.
Что содержит в себе транскрибация: каждое слово, а также невербальную коммуникацию (смех, паузы), звуки окружающей среды (кашель людей, плач детей, фоновый шум) и т.д.
Что не содержит в себе транскрибация: заминки и паузы, неуместные для транскрибации и затрудняющие чтение.
Примеры дословной транскрибации.
Понять особенности каждого из типов транскрибации можно из приводимых ниже примеров – отрывков из интервью с Джеффом Безосом (Jeff Bezos), президентом компании Amazon, транскрибированных с использованием каждого из типов транскрибации.
Транскрибация методом интеллектуальной дословной транскрибации:
«Ну, да, полагаю я не подумал об этом, верно. И потом, кроме того, у меня была куча ролевых моделей, которые я перепробовал в жизни. Некоторые из моих учителей в школе River Oaks Elementary, о которых вы упомянули.
У меня были мои родители, о которых я рассказывал немного ранее. Я не слишком много рассказывал о своем деде, но он оказал на меня большое влияние. Мне сильно повезло, поскольку моя мама была очень молода, каждое лето, начиная лет с четырех, я проводил у дедушки с бабушкой, чтобы мама немного отдохнула от меня – целое лето».
Дословная транскрибация:
«Ну, да, полагаю я не подумал об этом, верно. И потом, кроме того, у меня была куча ролевых моделей, которые я перепробовал в жизни. Да, некоторые из моих учителей в школе River Oaks Elementary, о которых вы упомянули. У меня были, у меня были мои родители, о которых я рассказывал немного ранее. Я не слишком много рассказывал о своем деде, но он оказал на меня большое влияние. Мне сильно повезло, поскольку моя мама была очень молода, каждое лето, даже начиная лет с четырех, я проводил у дедушки с бабушкой, просто чтобы мама немного отдохнула от меня – целое лето».
Настоящая дословная транскрибация:
«Ну, да, полагаю я не подумал об этом, ну, вы понимаете, верно.
Ммм…(сглатывает) и потом, кроме того, у меня была куча ролевых моделей, которые я перепробовал в жизни. Да, гм, (сглатывает) некоторые из моих учителей в школе River Oaks Elementary, о которых вы упомянули. У меня были, гм, у меня были мои родители, о которых я рассказывал немного ранее. Я не слишком много рассказывал о своем деде, но он оказал на меня большое влияние. Мне сильно повезло (сглатывает), поскольку моя мама была очень молода, каждое лето, даже… начиная лет с четырех, я проводил у дедушки с бабушкой, просто чтобы мама немного отдохнула от меня – целое лето».
Правила дословной транскрибации
Если вы только начинаете осваивать дословную транскрибацию, у вас могут появиться вопросы типа: «А правильно ли будет перефразировать некоторые фрагменты записи?» или: «А как мне транскрибировать смех на записи?».
Ниже приводятся четыре важных правила дословной и настоящей дословной транскрибации (в зависимости от того, насколько транскрибация подробна).
1. Записывайте каждое слово (не перефразируйте)
Многие из тех, кто занимается транскрибированием, зачастую перефразируют утверждения для передачи общего смысла того, что говорится, а не записывают речь слово в слово. Это по факту является интеллектуальной транскрибацией (см. описание типов выше).
Интеллектуальная транскрибация чаще всего используется в бизнесе (для расшифровки подкастов, совещаний, видео с You Tube и т.п.), поскольку подобные транскрибации легко читаются. Однако этот тип транскрибации не очень распространён в среде исследователей и журналистов, которым важна точность сказанного.
Ниже приводятся два примера, иллюстрирующих разницу между двумя стилями:
Перефразированное предложение: «Я кричал. Я звал свою мать. А она была, наверное, на расстоянии 30 ярдов от меня, в доме. Она бы не услышала меня, даже если бы была на улице».
Предложение, записанное дословно: «И я кричу, понимаете, я зову свою мать. А она была, хм, может быть в 30 ярдах от меня, в доме (пауза).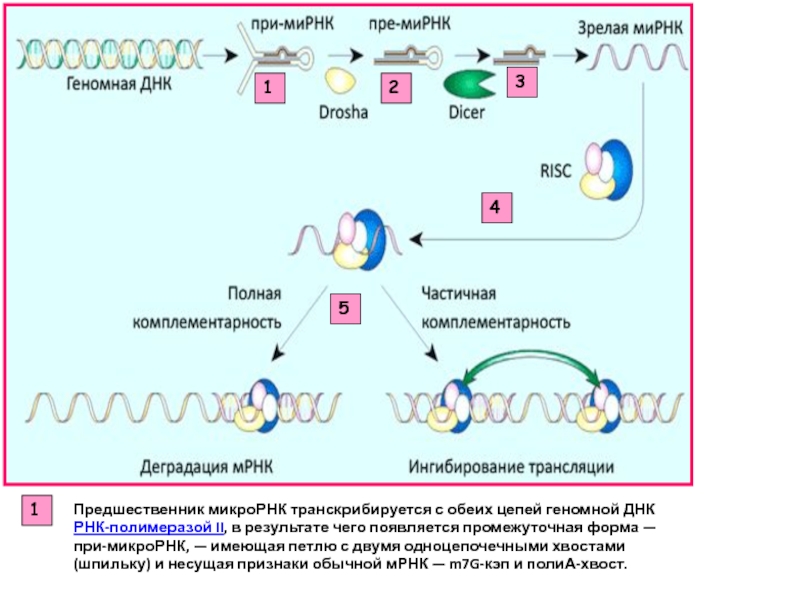 Я имею в виду…, она же не могла меня слышать. Даже если бы она была снаружи, она возможно не услышала бы меня».
Я имею в виду…, она же не могла меня слышать. Даже если бы она была снаружи, она возможно не услышала бы меня».
В то время как смысл в обеих версиях расшифровки не меняется, второй вариант значительно более эмоционален. В зависимости от того, для чего будет использоваться транскрибация, указанная разница может сыграть очень существенную роль.
Итак, в дословной транскрибации важно отражать каждое слово, которое произносится.
2. Не стоит игнорировать невербальную коммуникацию
Общение или коммуникация содержит в себе множество невербальных (несловесных) элементов, таких, как смех, паузы, звуки окружающей среды и прочее. Все эти элементы отражаются в дословной транскрибации.
Например, вот как смех зафиксирован в этом диалоге:
К.: Что думает твоя мама?
Н.: Ничего особенного. Она согласна со мной. Да.
К.: В самом деле? (смеется) Ты уверен?
(Н смеется)
Ещё некоторые правила для транскрибирования невербальной коммуникации.
Разговор, в котором реплики накладываются друг на друга.
Когда два собеседника говорят одновременно, в транскрибации это отражается знаком /, например:
Н.: Да, я тут/ жил.
К.: /Правда?
Н.: Три года.
Т.е. реплики «жил» и «правда» прозвучали одновременно и Н. продолжил свою фразу, не останавливаясь.
Используйте знак = когда, две строки следуют одна за другой без пробела, например:
К.: Она тебе понравилась? =
Н.: = Да!
Это был очень быстрый ответ.
Паузы.
Короткие паузы обозначайте точкой (.), многоточием (…) или словом (пауза).
К.: Поэтому, ретроспективно (пауза) это было плохое решение.
Если пауза длится более 4 секунд, указывайте продолжительность паузы в скобках, например (6) или (6 сек.).
Н.: Это было как (5 сек.) попасть со сковородки в огонь.
Прерывания.
Обозначайте прерывания коротким тире (-).
К.: Это было похоже на ц-
Н.: -Времени было так мало.
К. : Да.
: Да.
Смех.
Смех в транскрибации обозначается как (h) или (hhh) или (смех/смеется). (*Примечание: речь идет о транскрибации на английском языке)
Н.: Я до сих пор не верю, что нам это удалось (hhh).
3. Отражайте в транскрибации «холостые сообщения», заминки и неполные предложения
К холостым сообщениям и заминкам относятся междометия, фразы типа «вы понимаете», «вы знаете», часто используемые для того, чтобы выиграть время и подумать.
Неполные предложения, это предложения, которые остаются незаконченными, например:
«Я бы сказал, что не… я имею в виду, что это не может быть… лучше проконсультироваться со специалистом, прежде чем приступать к решению подобных вопросов».
Холостые сообщения и неполные предложения могут прерывать поток речи, но часто позволяют понять, о чем думает говорящий.
В дословную транскрибацию включаются все эти компоненты и не вымарываются при редактировании.
4. Фиксируйте внешние звуки
При проведении исследований качества, и даже маркетинговых исследований необходимо знать о том, что происходит вокруг, пока интервьюируемый говорит.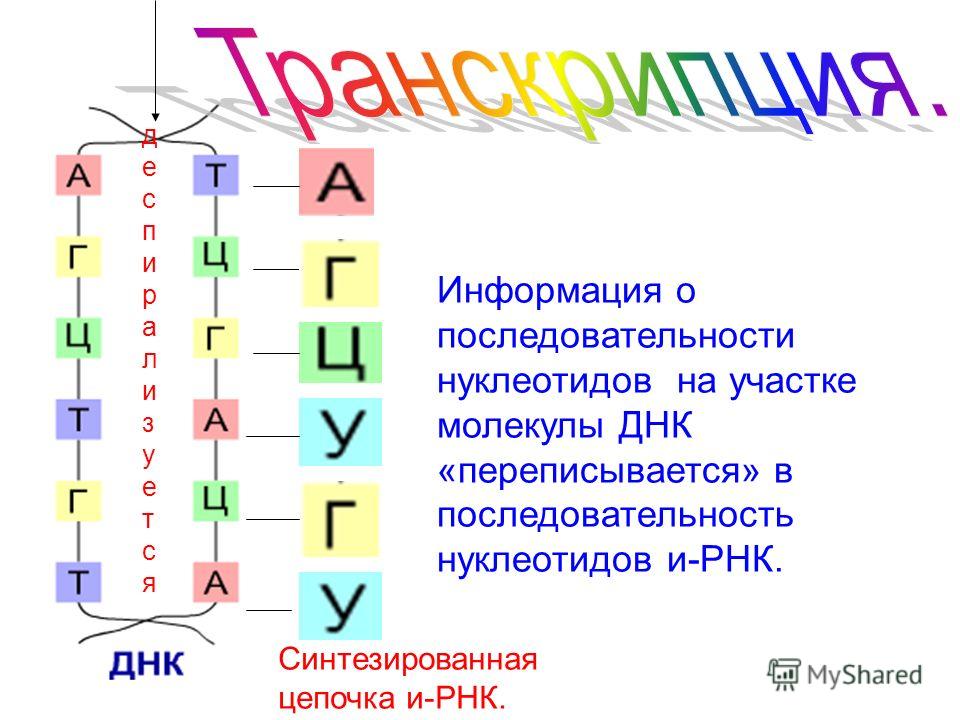
К внешним звукам можно отнести звук открываемой двери, шаги людей, фоновые чужие разговоры и пр. Эти звуки/события следует надлежащим образом отражать в транскрибации в скобках, отмечая время.
Основное назначение дословной транскрибации заключается в том, чтобы отражать как содержание, так и манеру речи.
Не всем нужны подробности – например, вам может потребоваться фиксация невербальной коммуникации, но при этом можно не фиксировать внешние звуки и события. Чтобы определиться с тем, что будет включено в расшифровку, а что нет, подумайте о том, как вы будете использовать транскрибацию.
Теперь, когда вы ознакомились с тем, что такое дословная транскрибация и каких типов она бывает, давайте рассмотрим некоторые общие для транскрибации вопросы.
Что такое удобочитаемая транскрибация?
Удобочитаемая транскрибация – это ещё одно название интеллектуальной транскрибации. Она называется удобочитаемой потому, что все неуместные слова, мешающие чтению, например, запинки, повторы и неполные предложения, из транскрибации вымарываются.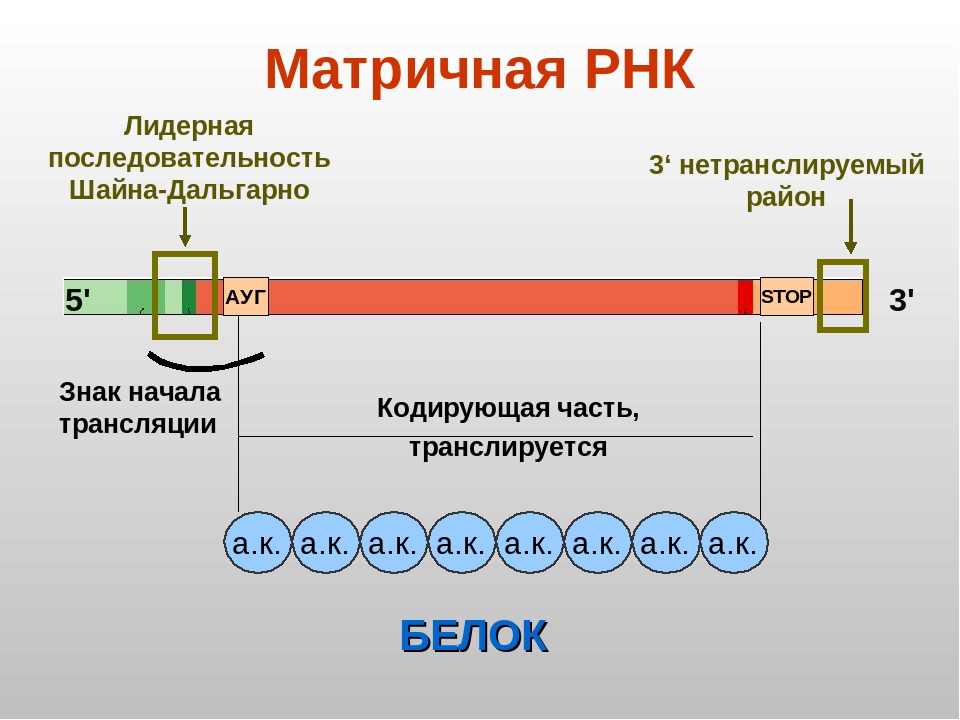 Вся остальная речь записывается в точности так, как она звучит на аудиозаписи.
Вся остальная речь записывается в точности так, как она звучит на аудиозаписи.
Что такое полная дословная транскрибация?
Полная дословная транскрибация – это ничто иное, как настоящая дословная транскрибация, в которой максимально подробно отражается каждое слово, каждый звук и невербальная коммуникация, присутствующая на аудиозаписи. Поскольку подобная транскрибация не редактируется, она называется полной.
Вот и всё, теперь вы знаете, что такое дословная транскрибация.
У вас появились вопросы? Пишите, и мы постараемся на них ответить.
Источник: https://www.indianscribes.com/4-rules-of-verbatim-transcription/
Специально для Бюро переводов СВАН Москва
Транскрипция и транскрибация в Москве
Постоянно развивающийся технический прогресс в свое время заменил живопись фотографией, прослушивание живой музыки магнитными, а позже и цифровыми записями. И даже письма, написанные от руки – уже редкость, затерявшаяся в миллионах «мейлов». Но в нашем динамичном мире не все получается заменить работой машин, аппаратов и компьютеров. Иногда человеческую работу не может выполнить никакой механизм. В частности воспринимать на слух речь и воплощать ее в текст на бумаге пока что не умеет ни одна машина. Поэтому транскрибация (а именно так и называется перевод аудио- или видео записей в текст) как вид работы пользуется сегодня большим спросом.
Что и зачем? Не спешите путать термин «транскрибация» с созвучной, например, «транскрипцией». Транскрипция – хоть и является также письменным воспроизведением слов, направлена на пояснение звучания каждого конкретного звука в слове. То есть задача транскрипции – обеспечить правильно чтение слова (зачастую, иностранного), передав графически запись его звучания для правильности произношения. А вот транскрибация – более обширный процесс, буквально означающий запись воспринимаемого на слух текста.
В последнее время транскрибация аудио в текст пользуется большой популярностью у людей, проводящих всевозможные семинары и вебинары. Удобно, правильно и эффективно, когда информация, выданная и полученная на них, имеет еще и текстовую интерпретацию. Кому-то это может понадобиться для научных работ, а кто-то воспринимает написанную или напечатанную информацию лучше, чем услышанную. С точки зрения контента информация, прошедшая такую обработку и воплотившаяся в текст, является уникальной, а значит и востребованной на просторах Интернета. Поэтому довольно часто на сайтах фрилансеров можно встретить предложения работы, в которых просят перевести в текстовый файл аудиозапись лекции, семинара, конференции и т.д.
Транскрибация аудио или видео файлов активно используется журналистами, в частности, когда они обрабатывают записанное в звуковом формате интервью. К ней прибегают авторы, когда начитывают свой текст на диктофон, а позже поручают сделать транскрибацию специалистам, чтобы получить непосредственно текст будущей книги.
Как?
Транскрибация имеет несколько видов:
- Дословный перевод текста со всеми звуками и междометиями говорящего
- Перевод с небольшими исправлениями – когда убирают повторяющиеся слова-паразиты, не несущие смысловую нагрузку связки и заменяют жаргон более литературными словами
- Литературный перевод – подразумевает стилистические исправления, вплоть до изменения предложений, их последовательности, оформление текста в разделы и подзаголовки.
Вакансия переводчика «из звука в текст» востребована, однако требует определенных навыков, умений и способностей.
- Прежде всего, нужно овладеть быстрым набором текста. И лучше вслепую.
- Уметь исправлять стилистические ошибки, в совершенстве владеть языком и, безусловно, быть грамотным с точки зрения орфографии и пунктуации.
- Воспринимать на слух значительные объемы информации, чтобы не делать слишком частых пауз для записи по три-четыре слова.
Как видите, труд весьма кропотливый. И выполнить его может только грамотный профессионал, обладающий знаниями. А пока не будет изобретена идеальная программа-анализатор для транскрибации, только человек сможет справиться с этой пусть и механической, но такой необходимой работой.
| Пользователь транскрибировал его, но я прокомментировал его / ее выступление, и было решено закрыть его, поэтому я не стал транскрибировать. | |
| Возможно ли для нас перейти на подстраницы и транскрибировать их вместо существующей системы, в которой каждый вопрос является просто разделом страницы? | |
| Чтобы начать транскрибировать ген, РНК-полимераза связывается с последовательностью ДНК, называемой промотором, и отделяет нити ДНК. | |
| Затем полимеразы могут считывать и” транскрибировать код РНК для синтеза мессенджерной РНК, или мРНК. | |
| РНК-полимераза может свободно продолжать транскрибировать весь оперон. | |
| Другие результаты | |
| Записная книжка зафиксировала, транскрибировала и перевела на японский язык путаные, невнятные объяснения. | |
| Когда пре-мРНК была транскрибирована из ДНК, она включает в себя несколько интронов и экзонов. | |
| Поэтому остальная часть оперона будет транскрибирована и переведена, так что триптофан может быть произведен. | |
| По состоянию на 2008 год она также разрабатывала несколько курсов для студентов в Беркли, в том числе один для изучения гармонии и теории через транскрибирование. | |
| Добросовестный перевод исходного материала на английский язык или транскрибирование произнесенных слов из аудио или видео источников не считается оригинальным исследованием. | |
| Псевдоген может быть получен, когда кодирующий ген накапливает мутации, которые препятствуют его транскрибированию, делая его нефункциональным. | |
| Это компания судебных стенографистов, нанятая для транскрибирования свидетельских показаний. | |
| Система счисления Нэшвилла — это метод транскрибирования музыки путем обозначения степени шкалы, на которой строится аккорд. | |
| Написание китайских географических или личных имен в пиньине стало самым распространенным способом их транскрибирования на английском языке. | |
| Входные последовательности для сборки EST являются фрагментами транскрибированной мРНК клетки и представляют собой только подмножество всего генома. | |
| Единственная роль наночастиц заключается в подавлении материнской транскрибированной горбатой мРНК в задней части тела. | |
| Редирект — это всего лишь ISBN; имена шаблонов, транскрибированные в статьях, должны давать некоторый намек на то, о чем они говорят. | |
| Редирект — это всего лишь ISBN; имена шаблонов, транскрибированные в статьях, должны давать некоторый намек на то, о чем они говорят. | |
| Редирект — это всего лишь ISBN; имена шаблонов, транскрибированные в статьях, должны давать некоторый намек на то, о чем они говорят. | |
| Ретротранспозоны могут быть транскрибированы в РНК, которые затем дублируются в другом месте генома. | |
| Это делает ДНК в хроматине доступной для транскрипционных факторов, позволяя генам транскрибироваться и экспрессироваться в клетке. | |
| У дрожжей и млекопитающих метилирование h4K36 сильно обогащено в организме высоко транскрибируемыми генами. | |
| Переход между средними бластулами также характеризуется заметным увеличением транскрипции новых, не материнских мРНК, транскрибируемых из генома организма. | |
| Гистон h4. 3 связан с организмом активно транскрибируемых генов. | |
| У млекопитающих Домены DNMT3a и DNMT3b PWWP связываются с h4K36me3, и эти два фермента рекрутируются в организм активно транскрибируемых генов. | |
| h4K27me1 связан с продвижением транскрипции и, как видно, накапливается в транскрибируемых генах. | |
| Как один из транскрибируемых генов, Wnts, секретируемые из эпителиальных клеток молочной железы, действуют паракринно, вызывая ветвление большего числа соседних клеток. | |
| Каждую неделю она транскрибирует интервью с Фэй, перерабатывает в главы книги и отправляет их мне на почту. | |
| На первом этапе сборки рибосомы белок под названием РНК-полимераза I транскрибирует рДНК, которая образует большой предшественник пре-рРНК. | |
| Когда РНК-полимераза связывает и транскрибирует ген trp, рибосома начинает переводить. | |
Лучшее приложение, которое, автоматически, транскрибирует аудио в текст
- Transcribe позволяет управлять воспроизведением звука, во время записи;
- Trint обеспечивает хорошую авто стенограмму;
- Descript позволяет обрезать аудио, редактируя текст.
Большинство авторов тратит много времени, на переписывание аудиозаписей в текст, который, затем, используется для статей. Мы не единственные, у кого возникает эта проблема: академики и исследователи, студенты и деловые люди, которые посещают множество встреч и нуждаются в том, чтобы транскрибировать аудио в текст и сэкономить время.
Наш нормальный рабочий процесс, для решения этой проблемы заключался в том, чтобы сохранить аудио файл, в QuickTime, в фоновом режиме, когда мы вводим текстовый редактор.
Есть несколько очевидных проблем с этим: например, такие вещи, как пауза и перемещение назад и вперед, излишне усложняются, когда вы перемещаетесь между программами, а во вторых — управлять скоростью воспроизведения, в соответствии со скоростью набора текста, тоже непросто. Короче говоря, это очень плохой рабочий процесс.
Приложение которое может решить эту проблему
В результате, мы, всегда, находимся в поиске хорошего приложения, которое может решить эту проблему, потому что это сделает жизнь намного проще. Во всяком случае, когда объем работы был слишком высоким, мы, на самом деле, прибегали к тому, чтобы получить кого-то из Freelancer.com, чтобы помочь записывать заметки о книгах, но это не самое хорошее решение, если у вас ограниченный бюджет.
Мы решили спросить людей, что они используют, и проверить технические сайты и форумы, такие как Product Hunt и Reddit, чтобы узнать, как найти лучшие варианты. Мы натолкнулись на множество рекомендаций, а затем, используя некоторые из наших интервью, взяли их, для пробных прогонов, чтобы увидеть, что может быть долгосрочным решением.
Потом, мы сузили все, до нескольких вариантов, которые, по нашему мнению, были самыми лучшими, а также, включают в себя некоторые очень разные типы решений. Существует три основных способа создания расшифровки стенограммы. Вы можете сделать это вручную, используя различные инструменты, которые делают процесс более эффективным.
Или вы, можете попытаться получить сгенерированную компьютером расшифровку, которая будет полна ошибок, но, по крайней мере, вы начнете процесс и тем самым сократите время, затрачиваемое на проект. Или вы, можете заплатить кому-то, чтобы транскрибировать стенограмму, как мы это делали с Freelancer.com. Мы сосредоточились на первых двух методах и вот наш лучший выбор.
Sonix
Приложение SonixSonix — это инструмент для транскрипции, основанный на веб-интерфейсах, который работал, достаточно хорошо. для нас. Мы попробовали сервис с четырьмя различными аудиоклипами и результаты были очень хорошими. Sonix поддерживает несколько языков, но кроме английского, маловероятно, что любой из них будет полезен. Sonix поддерживает американский, британский и австралийский акценты английского языка и имеет возможности, для всех других английских акцентов.
Мы загрузили четыре аудиоклипа на сайт, для тестирования Sonix.
Первым клипом, было интервью, с Томом Тейлором из Amazon, у которого был американский акцент. У этого клипа была самая лучшая скорость успешной транскрипции. Это было 30-минутное интервью, которое транскрибировалось, менее чем за 10 минут и текст, в целом, был неплохой.
Вторым клипом, было интервью, с основателем стартапа, в шумной обстановке и результаты были довольно плохими. Справедливости ради следует отметить, что Sonix упоминает, что для хорошего результата, требуется звук без фонового шума, но даже, без него, результаты были очень плохими.
Третий клип был четкой записью речи женщины, говорящей об инфраструктурной проблеме. Эта запись была переведена достаточно хорошо, за исключением некоторых слов, которые были неверными.
Заключительным клипом была запись телефонного разговора между двумя людьми, говорящими по-английски. Здесь не так много фонового шума и, первоначально, Sonix, полностью, перепутала транскрипцию. Мы предупредили компанию об этой проблеме и они ответили обновленной транскрипцией, которая была почти такой же точной, как и в третьем случае. Sonix заявляет, что это связано с несколькими системами транскрипции, которые у них есть и они использовали другую модель, для этой записи, когда мы предупреждали их о проблеме.
В нашем тестировании, Sonix оказался неплохим приложением, с высококачественными аудиофайлами, где оратор говорит в умеренных темпах. Когда в аудиозаписях говорят быстро, результаты Sonix были не такими уж хорошими. Тем не менее, приложение имеет несколько функций, которые позволяют проверить качество.
Плата за приложение Sonix
Нам очень понравился тот факт, что приложение имеет встроенный текстовый редактор, который позволяет быстро редактировать транскрипцию, во время прослушивания клипа. Скорость транскрипции, также, очень быстрая и наравне. с другими сервисами.
Если вы платите за услугу, приложение может различать два разных динамика и отмечать их. Лучшей особенностью, однако, является уверенный маркер, где приложение показывает, сколько слов, оно правильно расшифровало.
Sonix предлагает все эти функции и многое другое, за 6 долл. США в час, за записанные аудиофайлы, за исключением абонентской платы за подписку, в размере 15 долларов США, в месяц. Годовой план уменьшает цену, до 10 долл. США, в месяц. Цены не самые дешевые на рынке, но результаты с высококачественными записями достаточно хорошие, чтобы рассмотреть эту услугу.
Есть 30-минутная бесплатная, пробная версия, которую вы должны попробовать и увидеть результаты собственными глазами.
Transcribe
Transcribe — самая лучшая рекомендация, на разных платформах. Transcribe — это вариант, который нам, также, понравился, благодаря своей простоте и эффективности. Transcribe — это, в основном, аудиоплеер, с встроенным инструментом заметок, который позволяет слушать запись и делать заметки, в определенном месте.
Вы можете использовать сочетания клавиш, для ряда важных функций, связанных с воспроизведением и комбинация является серьезным шагом, от использования текстового редактора с QuickTime, в фоновом режиме.
Инструмент запускается на вашем компьютере в окне браузера, но он, также, работает в автономном режиме. Вы можете загружать аудио и сохранять текст локально, без каких-либо проблем. Звуковой файл воспроизводится с элементами управления, в верхней части страницы и у него есть текстовое поле ниже, где вы можете вводить текст, в комплекте с форматированием, а затем экспортировать его как DOC-файл, если это необходимо.
Ярлыки, с использованием функциональных клавиш, позволяют вам приостанавливать и воспроизводить, ускорять или замедлять звук, добавлять временную метку в текст и т. д. Если вы являетесь пользователем Mac, вам нужно перейти к настройкам и использовать клавиши, в качестве функциональных клавиш, а не управлять такими режимами, как яркость и громкость, а в остальном — это одно и то же.
Это, очевидно, лучшее решение, для нашего нормального рабочего процесса транскрипции и при использовании Transcribe, мы смогли конвертировать 30-минутную запись, в полезный текст, всего за 45 минут, то, что растягивалось для нас на час или, даже, дольше.
Также, есть интересное обходное решение, если вы хотите расшифровать, без ввода текста. Хотя, Transcribe не позволяет загружать аудиофайлы, вы можете диктовать слова и они будут, автоматически, вводится, если вы используете Chrome. Оно работает только в Chrome и поэтому, возможно, использование речи Google, для текстовых API, независимо от того, какой используется движок, результаты довольно точные, хотя, это не лучшее решение.
У нас, также, была проблема, когда курсор не двигался, последовательно, вперед. Несмотря на эти недостатки, если вы, некоторое время, используете функцию диктовки, вы привыкаете к ее причудам и это быстро и надежно.
Плата за приложение Transcribe
Транскрипция не является бесплатной и вы должны платить годовую лицензию, в размере 20 долл. США. Это очень хорошая сделка, если вы используете ее много, хотя, она может показаться немного дорогой, если вы, не используете ее часто.
Вы можете попробовать Transcribe, для себя, в течение недели и посмотреть, подходит ли вам это приложение.
Приложение TranscribeTrint
Trint — довольно простая услуга, которая автоматически транскрибирует аудиофайлы, которые вы загружаете и отправляет вам стенограмму. Trint позволяет вам загружать файл, а затем транскрибировать его в интернете. Когда это будет сделано (что зависит от длины аудиофайла), вы получите электронное письмо, уведомляющее вас, чтобы вы могли закрыть окно и выполнить другую работу. Это не занимает много времени — транскрибирование 10-минутного файла занимает около четырех минут.
Однако, Trint не только предоставляет текстовый файл. Кроме этого, после расшифровки, он предоставляет мощный текстовый редактор, который позволяет прослушивать воспроизведение, при редактировании текста, так же как и Transcribe.
Вы даже, можете пометить различные разделы текста динамиком или добавить подсветку. Когда вы закончите, вы можете экспортировать текст, который может быть как .DOC-файл, или файл субтитров .SRT или если, вам нужны только части файла, вы можете выбрать экспорт, только основных моментов.
Вы можете изменить скорость воспроизведения, показать временную метку, для каждого абзаца или перемещаться по тексту, перемещаясь назад и вперед, через аудиофайл. По мере воспроизведения звука, соответствующий текст, также, выделяется, поэтому его очень легко отслеживать.
Это очень здорово, хотя, одно ограничение заключается в том, что вы можете использовать его, только, на своем компьютере, потому что приложений для iOS и Android нет.
Точность транскрипции, также, оставляет желать лучшего. По большому счету, текст довольно чистый, а его правильность составляет около 70 процентов.
Плата за приложение Trint
С вас будет взиматься плата, в размере 15 долл. США в час, что не так уж и плохо, потому что запись и транскрипция (со всеми внесенными вами изменениями) всегда доступны, когда вам это нужно. Вы можете попробовать Trint, в течение 30 минут, бесплатно и посмотреть, насколько он подходит вашим потребностям.
Scribie
Если вы не заинтересованы в оплате, вы, также, можете использовать Scribie, которая предлагает неограниченную бесплатную машинную транскрипцию.
Scribie, менее точное приложение и делает лучше всего транскрипцию, с очень четким звуком.
В Scribie, также, есть транскрипт, обработанный человеком, для которого взимается плата, в размере 0,60 доллара США в минуту, в течение, максимум, пяти дней.
Descript
Если вам понравилась идея Trint, но думаете, что интерфейс оставляет желать лучшего и ему не понравилась идея запуска приложения, в вашем браузере, тогда выбирайте Descript, вместо него. Приложение бесплатное и поставляется с 30-минутной бесплатной транскрипцией, после чего, вы будете платить 0,15 доллара США в минуту, что довольно разумно.
Приложение Descript 1.0, для Windows.В Descript есть великолепное приложение, для Mac, которое позволяет вам делать все, что делает Trint, начиная с автоматической транскрипции, а затем, позволяя редактировать текст.
Вы можете пометить текст, чтобы пропустить воспроизведение звука, исправить ошибки и создать плавный скрипт, который, идеально, соответствует звуку. Это, действительно, здорово и обладает всеми функциями, которые вам нужны в интерфейсе, который нам понравился.
Когда вы перемещаете текст, приложение, также, показывает ваше место в аудиофайле и позволяет публиковать отредактированный звук и текст в интернете, если вы, хотите этого. Приложение работает от Google Speech и оно довольно точное, хотя, очевидно, есть некоторые ошибки. Мы обнаружили, что оно, почти на 80 процентов точное, если звук был четким, без помех.
Плата за приложение Descript
В описании, также, предлагается ежемесячный абонентский план, в котором вы платите 20 долларов, в месяц, но тогда, ваша плата за минуту, составит 0,07 доллара, что является хорошим вариантом, для постоянных пользователей.
Вы можете, бесплатно, загрузить Descript и попробовать его, в течение 30-минутного файла, чтобы понять, как он работает, прежде чем платить или подписываться на подписку. Версия, для Windows, появилась, в январе 2018 года. Для Descript, также, нет мобильной версии.
Заключение
По нашему опыту, Descript, вероятно, был лучшим инструментом, из этой группы, хотя, ее цена, за минуту, не совсем приемлема. На данный момент, мы склонны к Transcribe, поскольку оно предлагает годовую подписку, без каких-либо дополнительных затрат, а режим диктовки — это шаг вперед, от Transcribe.
Были, также, несколько мобильных приложений, которые обещали аналогичный опыт, но в нашем тестировании были ограничены. Транскрибирование, которое включает в себя, достаточное количество ввода, на сенсорном экране, по-прежнему, оставляет желать лучшего и лучше всего придерживаться этих опций, на базе ПК.
Смотрите также:
Транскрибироваться — понятие и значение
Рассмотрим что означает понятие и значение слова транскрибироваться .
Транскрибироваться это — 1. Не совершенный вид
2. Поддаваться транскрипции.
3. Страд. к несовершенный вид глагол : транскрибировать.
Транскрибироваться это — 1. Транскрибируюсь, транскрибируешься, несовершенный вид ( специальное ). 1. Страд. к транскрибировать. 2. Быть изображаемым тем или иным способом в транскрипции (о словах, звуках речи). Английское «и» транскрибируется по-русски то через «у», то через «э», то через «а».
-руется; несовершенный вид Лингв.
1.
Быть изображаемым, передаваемым тем или иным способом транскрипции.
2.
Страд. к транскрибировать.
Часть речи
Глагол (инфинитив)
См. также
… места ударения (‘перед ударным слогом ). при фонематической транскрипции ударение не обозначается , а транскрибированные морфемы соединяются дефисами в пределах слов , отделяемых пробелами . образец фонетической и фонематической … (ТЕОРИЯ ПЕРЕВОДА)
… интерфейса можно диктовать запросы в клиентской программе на устройстве , а не вводить их Чтобы транскрибировать продиктованы слова в написанный текст , Google направляет выражения серверы , где используется технология … (Распознавание образов)
… »; , « защита от осадков »; и «навыки выживания в ядерной войне »; , также были обычным явлением . В транскрибированной радиопрограмме « звезды обороны »; хитовая музыка сочетается с советами гражданской обороны . государственные … (Безопасность жизнедеятельности)
транскрибировать
транскрипционный
Транскрипция
РНК | Микробиология
Цели обучения
- Объясните, как синтезируется РНК с использованием ДНК в качестве матрицы
- Различать транскрипцию у прокариот и эукариот
В процессе транскрипции информация, закодированная в последовательности ДНК одного или нескольких генов, транскрибируется в цепь РНК, также называемую транскриптом РНК . Полученная одноцепочечная молекула РНК, состоящая из рибонуклеотидов, содержащих основания аденин (A), цитозин (C), гуанин (G) и урацил (U), действует как мобильная молекулярная копия исходной последовательности ДНК.Транскрипция у прокариот и эукариот требует, чтобы двойная спираль ДНК частично раскручивалась в области синтеза РНК. Развернутая область называется пузырем транскрипции . Транскрипция определенного гена всегда происходит от одной из двух цепей ДНК, которая действует как матрица, так называемой антисмысловой цепи . Продукт РНК комплементарен матричной цепи ДНК и почти идентичен нетемплатной цепи ДНК или смысловой цепи .Единственное отличие состоит в том, что в РНК все нуклеотиды Т заменены на нуклеотиды U; во время синтеза РНК U включается, когда в комплементарной антисмысловой цепи присутствует A.
Транскрипция бактерий
Бактерии используют одну и ту же РНК-полимеразу для транскрипции всех своих генов. Подобно ДНК-полимеразе, РНК-полимераза добавляет нуклеотиды один за другим к 3′-ОН группе растущей нуклеотидной цепи. Одним из критических различий в активности между ДНК-полимеразой и РНК-полимеразой является потребность в 3′-ОН, к которому можно добавить нуклеотиды: ДНК-полимеразе требуется такая 3′-ОН группа, что требует праймера, тогда как РНК-полимеразы нет.Во время транскрипции рибонуклеотид, комплементарный матричной цепи ДНК, добавляется к растущей цепи РНК, и ковалентная фосфодиэфирная связь образуется посредством синтеза дегидратации между новым нуклеотидом и последним добавленным нуклеотидом. В E. coli РНК-полимераза состоит из шести полипептидных субъединиц, пять из которых составляют основной фермент полимеразы, ответственный за добавление нуклеотидов РНК к растущей цепи. Шестая субъединица известна как сигма (σ). σ-фактор позволяет РНК-полимеразе связываться со специфическим промотором, тем самым обеспечивая транскрипцию различных генов.Существуют различные σ-факторы, которые позволяют транскрипцию различных генов.
Инициирование
Инициирование транскрипции начинается с промотора , последовательности ДНК, с которой связывается аппарат транскрипции и инициирует транскрипцию. Нуклеотидная пара в двойной спирали ДНК, которая соответствует сайту, из которого транскрибируется первый 5′-нуклеотид РНК, является сайтом инициации. Нуклеотиды, предшествующие сайту инициации, обозначены как «вышестоящие», тогда как нуклеотиды, следующие за сайтом инициации, называются «нижележащими» нуклеотидами.В большинстве случаев промоторы расположены непосредственно перед генами, которые они регулируют. Хотя промоторные последовательности различаются в зависимости от бактериального генома, некоторые элементы сохраняются. В положениях –10 и –35 внутри ДНК перед сайтом инициации (обозначенным +1) есть две консенсусные последовательности промоторов или области, которые схожи для всех промоторов и у разных видов бактерий. Консенсусная последовательность –10, называемая блоком TATA , является TATAAT. Последовательность –35 распознается и связывается с σ.
Удлинение
Элонгация в фазе транскрипции начинается, когда σ-субъединица диссоциирует от полимеразы, позволяя коровому ферменту синтезировать РНК, комплементарную матрице ДНК, в направлении от 5 ‘к 3’ со скоростью примерно 40 нуклеотидов в секунду. По мере удлинения ДНК непрерывно разматывается перед основным ферментом и снова наматывается за ним (рис. 1).
Рис. 1. Во время элонгации бактериальная РНК-полимераза отслеживает ДНК-матрицу, синтезирует мРНК в направлении от 5 ‘к 3’ и раскручивает и перематывает ДНК по мере ее считывания.
Прекращение действия
После того, как ген транскрибируется, бактериальная полимераза должна отделиться от матрицы ДНК и высвободить вновь образованную РНК. Это называется прекращением транскрипции . Матрица ДНК включает повторяющиеся нуклеотидные последовательности, которые действуют как сигналы терминации, заставляя РНК-полимеразу останавливаться и высвобождаться из матрицы ДНК, высвобождая транскрипт РНК.
Подумай об этом
- Где σ-фактор РНК-полимеразы связывает ДНК, чтобы начать транскрипцию?
- Что происходит, чтобы инициировать полимеризационную активность РНК-полимеразы?
- Откуда исходит сигнал об окончании транскрипции?
Транскрипция в эукариотах
Прокариоты и эукариоты выполняют в основном одинаковый процесс транскрипции с некоторыми существенными различиями (см. Таблицу 1).Эукариоты используют три различных полимеразы, РНК-полимеразы I, II и III, все структурно отличные от бактериальной РНК-полимеразы . Каждый транскрибирует разные подмножества генов. Интересно, что архей содержат единственную РНК-полимеразу, которая более тесно связана с эукариотической РНК-полимеразой II, чем с ее бактериальным аналогом. Эукариотические мРНК также обычно являются моноцистронными, что означает, что каждая из них кодирует только один полипептид, тогда как прокариотические мРНК бактерий и архей обычно являются полицистронными , что означает, что они кодируют несколько полипептидов.
Наиболее важным различием между прокариотами и эукариотами является мембраносвязанное ядро последних, которое влияет на простоту использования молекул РНК для синтеза белка. Когда гены связаны в ядре, эукариотическая клетка должна транспортировать кодирующие белок молекулы РНК в цитоплазму для трансляции. Кодирующие белок первичные транскрипты , молекулы РНК, непосредственно синтезируемые РНК-полимеразой, должны пройти несколько этапов процессинга, чтобы защитить эти молекулы РНК от деградации в течение времени, когда они переносятся из ядра в цитоплазму и транслируются в белок.Например, мРНК эукариот может длиться несколько часов, тогда как типичная мРНК прокариот длится не более 5 секунд.
Первичный транскрипт (также называемый пре-мРНК) сначала покрывается РНК-стабилизирующими белками, чтобы защитить его от деградации, пока он обрабатывается и экспортируется из ядра. Обработка первого типа начинается, пока синтезируется первичная расшифровка; специальный 7-метилгуанозиновый нуклеотид, называемый 5′-кэп , добавляется к 5′-концу растущего транскрипта.Помимо предотвращения деградации, факторы, участвующие в последующем синтезе белка, распознают кэп, что помогает инициировать трансляцию рибосомами. После завершения удлинения другой процессирующий фермент затем добавляет цепочку из примерно 200 адениновых нуклеотидов к 3′-концу, называемую хвостом поли-А . Эта модификация дополнительно защищает пре-мРНК от деградации и сигналов клеточным факторам о том, что транскрипт необходимо экспортировать в цитоплазму.
Эукариотические гены, кодирующие полипептиды, состоят из кодирующих последовательностей, называемых экзонами ( ex -on означает, что они сжаты ex ) и промежуточных последовательностей, называемых интронами ( int -ron обозначает их исходную роль int ).Транскрибируемые последовательности РНК, соответствующие интронам, не кодируют участки функционального полипептида и удаляются из пре-мРНК во время процессинга. Важно, чтобы все последовательности РНК, кодируемые интроном, были полностью и точно удалены из пре-мРНК перед синтезом белка, чтобы последовательности, кодируемые экзонами, были правильно соединены вместе для кодирования функционального полипептида. Если процесс ошибается даже на один нуклеотид, последовательности соединенных экзонов будут сдвинуты, и полученный полипептид окажется нефункциональным.Процесс удаления последовательностей РНК, кодируемых интроном, и воссоединения последовательностей, кодируемых экзонами, называется сплайсингом РНК и облегчается действием сплайсосомы , содержащей малые ядерные рибонуклеопротеины (мяРНП). Последовательности РНК, кодируемые интроном, удаляются из пре-мРНК, пока она еще находится в ядре. Хотя они не транслируются, интроны, по-видимому, выполняют различные функции, включая регуляцию генов и транспорт мРНК. По завершении этих модификаций зрелый транскрипт , мРНК, кодирующая полипептид, транспортируется из ядра и направляется в цитоплазму для трансляции.Интроны можно сплайсировать по-разному, в результате чего различные экзоны включаются или исключаются из конечного продукта мРНК. Этот процесс известен как альтернативное сращивание . Преимущество альтернативного сплайсинга заключается в том, что могут быть созданы различные типы транскриптов мРНК, все из которых происходят из одной и той же последовательности ДНК. В последние годы было показано, что некоторые археи также обладают способностью сплайсировать свою пре-мРНК.
| Таблица 1. Сравнение транскрипции у бактерий и эукариот | ||
|---|---|---|
| Имущество | Бактерии | Эукариоты |
| Количество полипептидов, кодируемых на мРНК | Моноцистронный или полицистронный | Исключительно моноцистроник |
| Удлинение прядей | ядро + σ = холоэнзим | РНК-полимеразы I, II или III |
| Добавление 5-дюймовой крышки | № | Есть |
| Добавление 3 ‘хвоста поли-А | № | Есть |
| Сплайсинг пре-мРНК | № | Есть |
Визуализируйте, как происходит сплайсинг мРНК, наблюдая за процессом в этом видео.
Посмотрите, как удаляются интроны во время сплайсинга РНК.
Подумай об этом
- В эукариотических клетках, как транскрипт РНК из гена белка изменяется после его транскрипции?
- Содержат ли экзоны или интроны информацию о белковых последовательностях?
Клиническая направленность: Трэвис, часть 2
Этот пример продолжает историю Трэвиса, начатую в книге «Функции генетического материала».
В отделении неотложной помощи медсестра сказала Трэвису, что он принял правильное решение поехать в больницу, потому что его симптомы указывали на инфекцию, которая вышла из-под контроля.Симптомы Трэвиса прогрессировали, пораженный участок кожи и опухоль увеличивались. Внутри пораженного участка появилась сыпь, образовались пузыри и небольшие газовые карманы под самым внешним слоем кожи, и некоторые участки кожи стали серыми. Учитывая гнилостный запах гноя, вытекающего из одного из пузырей, быстрое прогрессирование инфекции и внешний вид пораженной кожи, врач немедленно начал лечение некротического фасциита.Врач Трэвиса назначил посев жидкости, вытекающей из волдыря, а также назначил анализ крови, в том числе определение количества лейкоцитов.
Трэвис был помещен в отделение интенсивной терапии и начал внутривенное введение антибиотика широкого спектра действия, чтобы попытаться минимизировать дальнейшее распространение инфекции. Несмотря на лечение антибиотиками, состояние Трэвиса быстро ухудшилось. Трэвис смутился, и у него закружилась голова. В течение нескольких часов после госпитализации его кровяное давление значительно упало, а дыхание стало более поверхностным и учащенным.Вдобавок волдыри увеличивались, волдыри становились пурпурно-черными, а сама рана, казалось, быстро прогрессировала вверх по ноге Трэвиса.
- Какие возможные возбудители некротического фасциита Трэвиса?
- Каковы возможные объяснения того, почему лечение антибиотиками не работает?
Мы вернемся к примеру Трэвиса на следующих страницах.
Ключевые концепции и резюме
- Во время транскрипции информация, закодированная в ДНК, используется для создания РНК.
- РНК-полимераза синтезирует РНК, используя антисмысловую цепь ДНК в качестве матрицы путем добавления нуклеотидов комплементарной РНК к 3′-концу растущей цепи. РНК-полимераза
- связывается с ДНК в последовательности, называемой промотором , во время инициации транскрипции .
- Гены, кодирующие белки родственных функций, часто транскрибируются под контролем одного промотора в прокариотах, что приводит к образованию полицистронной молекулы мРНК , которая кодирует несколько полипептидов.
- В отличие от ДНК-полимеразы, РНК-полимераза не требует 3′-OH-группы для добавления нуклеотидов, поэтому праймер не требуется во время инициации.
- Прекращение транскрипции у бактерий происходит, когда РНК-полимераза сталкивается со специфическими последовательностями ДНК, которые приводят к остановке полимеразы. Это приводит к высвобождению РНК-полимеразы из цепи ДНК-матрицы, высвобождая транскрипт РНК .
- Эукариоты имеют три различных РНК-полимеразы.У эукариот также есть моноцистронная мРНК, каждая из которых кодирует только один полипептид.
- Первичные транскрипты эукариот обрабатываются несколькими способами, включая добавление 5′-кэпа и 3′- поли-A-хвоста , а также сплайсинг для создания зрелой молекулы мРНК, которая может быть транспортирована. ядра, и это защищено от деградации.
Множественный выбор
На какой стадии бактериальной транскрипции участвует σ-субъединица РНК-полимеразы?
- инициирование
- удлинение
- прекращение
- сварка
Ответ а.Субъединица σ РНК-полимеразы участвует в инициации.
Какой из следующих компонентов участвует в инициации транскрипции?
- грунтовка
- происхождение
- промоутер
- стартовый кодон
Ответ c. Промотор участвует в инициации транскрипции.
Что из следующего не является функцией 5′-кэпа и 3′-поли-А-хвоста молекулы зрелой эукариотической мРНК?
- для облегчения сварки
- для предотвращения деградации мРНК
- для экспорта зрелого транскрипта в цитоплазму
- для облегчения связывания рибосомы с транскриптом
Ответ а.Облегчение сращивания не зависит от 5′-колпачка и 3′-хвоста поли-А.
Зрелая мРНК эукариота будет содержать каждую из этих особенностей, кроме какой из следующих?
- экзон-кодируемая РНК
- РНК, кодируемая интроном
- 5 ‘крышка
- 3 ′ хвостовик поли-А
Ответ б. Зрелая мРНК эукариота будет , а не , содержать РНК, кодируемую интроном.
Заполните бланк
A ________ мРНК — это мРНК, которая кодирует несколько полипептидов.
Показать ответПолицистронная мРНК — это мРНК, кодирующая несколько полипептидов.
Белковый комплекс, отвечающий за удаление кодируемых интроном последовательностей РНК из первичных транскриптов у эукариот, называется ________.
Показать ответБелковый комплекс, ответственный за удаление кодируемых интроном последовательностей РНК из первичных транскриптов у эукариот, называется сплайсосомой .
Подумай об этом
- Какова цель процессинга РНК у эукариот? Почему прокариотам не нужна подобная обработка?
- Ниже представлена последовательность ДНК.Представьте себе, что это часть молекулы ДНК, которая отделилась при подготовке к транскрипции, поэтому вы видите только антисмысловую цепь. Постройте последовательность мРНК, транскрибируемую с этой матрицы. Антисмысловая цепь ДНК: 3′-T A C T G A C T G A C G A T C-5 ‘
- Предсказать эффект изменения последовательности нуклеотидов в области –35 бактериального промотора.
Инициирование транскрипции РНК-полимеразы II: структурный вид
У эукариот РНК-полимераза II транскрибирует информационные РНК и несколько малых ядерных РНК.Как и РНК-полимеразы I и III, полимераза Я-я не могу действовать в одиночку. Вместо этого общие факторы инициации [фактор транскрипции (TF) IIB, TFIID, TFIIE, TFIIF и TFIIH] собираются на промоторной ДНК с помощью полимеразы II, создавая большой комплекс мультипротеин-ДНК, который поддерживает точное инициирование. Другой группа акцессорных факторов, активаторов транскрипции и коактиваторы, регулируют скорость синтеза РНК из каждого гена в реакция на различные сигналы развития и окружающей среды. Наш обзор текущих знаний об этом сложном макромолекулярном механизме подробно, с особым акцентом на идеи, полученные из структурных исследования факторов транскрипции.
Эукариотическая РНК-полимераза II (pol II) представляет собой 12-субъединицу ДНК-зависимая РНК-полимераза, отвечающая за транскрипцию ядерные гены, кодирующие информационные РНК и несколько малых ядерных РНК (1). Несмотря на очевидную структурную сложность, эта мультисубъединица ферменту требуются две группы вспомогательных белков для решения двух критических биохимические проблемы. Во-первых, pol II не может распознать свою цель промоутеры напрямую. Во-вторых, pol II должен уметь модулировать производство транскриптов РНК отдельных генов в ответ на онтогенетические и сигналы окружающей среды.
Анатомия промотора и преинициативный комплекс (PIC)
Промоторы ядерных генов класса II содержат комбинации ДНК. последовательности, которые включают основные или базальные элементы промотора, промотор проксимальные элементы и дистальные энхансерные элементы. Транскрипция инициация pol II точно регулируется факторами транскрипции (белки), которые взаимодействуют с этими тремя классами ДНК-мишеней и также друг с другом (см. ссылки 2–4). Лучше всего охарактеризован основные промоторные элементы, которые могут функционировать независимо или синергетически это элемент ТАТА (расположенный на 25 п.н. выше сайт начала транскрипции с консенсусной последовательностью TATAa / tAa / t), и богатый пиримидином инициаторный элемент (расположенный в стартовом сайте).В core-промотор представляет собой основную ДНК-мишень для pol II, и точная инициация транскрипции зависит от сборки pol II и факторы транскрипции (TF) IID, IIB, IIF, IIE и IIH в PIC (Таблица 1; Рис. 1). это считали, что эти факторы транскрипции необходимы для позиционирования pol II на большинстве промоторов ядерных генов класса II, и они обычно называемые общими факторами инициирования (см. ссылку 2). Таким образом, PIC функционально эквивалентен гораздо более простому Escherichia coli холоэнзим, который состоит из ядра Субъединицы РНК-полимеразы и σ-фактор (см.6). Промоутер проксимальные элементы расположены где-то между 50 и 200 п.н. перед стартовый сайт и связывание активаторов транскрипции с этими последовательностями регулируют транскрипцию. Наконец, дистальные энхансерные элементы, которые могут быть находится далеко от сайта инициации транскрипции в любом направлении и ориентации, составляют еще одну группу ДНК-мишеней для факторов модулирующий активность pol II.
Факторы инициации транскрипции общего класса II из клетки человека
( A ) Сборка PIC начинается с TFIID распознавание элемента ТАТА с последующим скоординированным наращиванием TFIIB, нефосфорилированная форма pol II (pol IIA) плюс TFIIF, TFIIE и TFIIH.Перед удлинением pol II фосфорилируется (pol IIO). После завершения фосфатаза рециклирует pol II в свой нефосфорилированная форма, позволяющая ферменту повторно инициировать транскрипцию in vitro . Привязка TBP (и TFIID) к блоку TATA является по сути медленный шаг, приводящий к долгоживущему комплексу белок-ДНК. Эффективная реинициация транскрипции может быть достигнута, если переработанный pol II повторно входит в преинициативный комплекс до того, как TFIID диссоциирует от основной промоутер. (По материалам исх.5.) ( B ) Схема представление функциональных взаимодействий, которые модулируют базальную ( Upper ) и активатор-зависимая транскрипция ( Нижний ). Базальные факторы TBP, TFIIB, TFIIF, TFIIE и TFIIH и pol II обозначены желтыми символами с общим содержание фактора инициации «холофермента pol II», заключенного в квадратных скобок. TAF II и без TAF II показаны коактиваторы (фиолетовый) и активаторы транскрипции (зеленый) взаимодействуют со своими целями в ПОС.(Изображение предоставлено R.G. Рёдер и С. Стивенс, Университет Рокфеллера.)
Фактор транскрипции IID
В самом общем случае производство матричной РНК начинается с TFIID распознает и жестко связывается с элементом TATA (рис. 1). Важная роль TFIID сделала его центром значительного биохимические и генетические исследования с момента его открытия на клетках человека в 1980 (7). Наша текущая перепись клонированных субъединиц TFIID включает больше более дюжины различных полипептидов массой от 15 до 250 кДа (рассмотрено в исх.8). Большинство этих субъединиц TFIID отображают значительная консервация среди человека, Drosophila и дрожжей, подразумевая общий наследственный TFIID, и исследования нарушения генов четырех субъединицы TFIID дрожжей показали, что они необходимы для жизнеспособности (9, 10).
Связывание ДНКчеловеческим TFIID было впервые продемонстрировано с аденовирусом. основной поздний промотор (AdMLP) (11). Исследования футпринтинга ДНКазы I AdMLP и промоторы отдельных генов человека выявили специфичные для последовательности взаимодействия между человеческим TFIID и элементом TATA, которые в первую очередь опосредуется субъединицей TBP TFIID (см. ниже).В Напротив, защита как перед, так и после элемента TATA в значительной степени независима от последовательности, демонстрирует нуклеосомоподобный образец Гиперчувствительность к ДНКазе I, радикально варьирует между промоторами и может индуцироваться некоторыми активаторами (см. ссылку 8). Это замечательно что связывание коробки TATA с помощью TFIID или TBP препятствует упаковке коровой промотор с несвязными гистоновыми белками (h3A, h3B, h4 и h5). Напротив, упаковка ядерного промотора октамерами гистонов в нуклеосомы предотвращают связывание TFIID или TBP с элементом TATA, эффективно подавляя транскрипцию (см.12). В vivo , репрессия транскрипции, опосредованная хроматином, преодолевается различные АТФ-зависимые макромолекулярные машины (например, SWI / SNF комплекс), который ремоделирует хроматин в непосредственной близости от корового промотора (рассмотрено в ссылке 13).
ТАТА-бокс-связывающий белок
После публикации последовательности дрожжевого ТВР в 1989 г. быстро последовательностями гомологичных генов от различных эукариот и архебактерии (идентичность аминокислот в пределах филогенетически консервативная часть из 180 остатков составляет от 38% до 100%, рассмотрено в исх.14). Один рекомбинантный ТВР может связывать оба общие и нормативные факторы и прямая сборка ПОС в vitro и базальная транскрипция (см. ссылку 2). Базальный или стержневой промотор-зависимая транскрипция является относительно неэффективной в vitro , которая послужила важным инструментом для характеризуя минимальные требования Pol II к TBP, TFIIB, TFIIF, TFIIE, TFIIH и центральная область промотора сразу перед сайтом начала транскрипции. Помимо определения минимальные факторы, необходимые для точного инициирования Pol II, базальная система транскрипции была использована для установления порядка PIC сборка, изображенная на рис.1 А .
Рис. 1 B контрастирует базальная и активированная транскрипция. (рассмотрено в ссылке 8). Активированная транскрипция in vivo требуется весь промотор, который включает центральную область плюс проксимальная и дистальная области энхансера промотора. Транскрипционный активаторы регулируют эффективность инициации pol II, распознавая их последовательности ДНК-мишени в проксимальном или дистальном энхансере промотора элементы и взаимодействие с PIC (возможно, через посредников известные как коактиваторы).Для активированной транскрипции требуется TBP и остальные субъединицы TFIID ( TAF II s), другие общие факторы инициации TFIIB, TFIIF, TFIIE и TFIIH, а также активаторы и коактиваторы транскрипции.
Трехмерная структура консервативной части ТВР поразительно похожий на седло (14–16) (рис. 2 A ), что идеально соответствует Биохимическая функция ТВР как белка, который сидит на ДНК, создавая стабильная платформа для связывания других факторов транскрипции.Связывание ДНК поддерживается вогнутой нижней стороной седла, в то время как выпуклая верхняя поверхность или посадочное место седла связывает различные компоненты аппарат транскрипции (см. ссылку 14). ТБП состоит из двух квазиидентичные домены (рис.2 A ), соответствующие два прямых повтора, обнаруженные в консервативной части ТВР (15). Следовательно, предок ТВР мог функционировать как димер с геном дупликация и слияние, приводящее к мономерному квазисимметричному ТВР.
( A ) Трехмерные конструкции TBP (14–16) ( верхний левый ), TBP в комплексе с TATA элемент (17–19) ( верхний правый ), клемма C или сердцевина TFIIB (cTFIIB) –TBP – TATA элементный тройной комплекс (20) ( Нижний Слева ) и тройной элемент TFIIA – TBP – TATA (21, 22) ( Нижний правый ).Белки изображены в виде ленты. чертежи, с маркированными концами N и C, если они видны. ДНК показаны в виде фигурки с гипотетическими линейными удлинениями B-формы на обоих концах. Сайт начала транскрипции AdMLP помечен +1. TBP, а комплексы TBP – ДНК и cTFIIB – TBP – ДНК показаны из та же самая выгодная точка после сайта начала транскрипции. В Комплекс TFIIA – TBP – ДНК рассматривается выше элемента TATA, смотрит в сторону начала транскрипции.Молекулы имеют цветовую маркировку следующим образом: красный, первый повтор cTFIIB; пурпурный, второй повтор cTFIIB; светло-голубой, конец TBP N и первый повтор; темно-синий, секунда TBP повторить; зеленый — малая субъединица TFIIA; желтый — большая субъединица TFIIA; а также серый, ДНК. Когда TBP распознает малую бороздку элемента TATA, ДНК изгибается и разматывается, чтобы представить края малых бороздок основания к нижней стороне молекулярного седла. На cTFIIB или TFIIA связывания с комплексом ТВР-ДНК практически нет изменений в структура бинарного комплекса.( B ) Детали конструкции TFIIB. Относительная ориентация двух доменов cTFIIB в свободная и связанная форма совершенно разные. Связанные и свободные cTFIIB нарисованы с выровненными их первыми доменами. Концы N и C фрагменты белка, использованные в структурных исследованиях, помечены, а α-спирали каждого домена cTFIIB окрашены в красный, зеленый, синий, желтый и пурпурный. Спираль присутствует только во второй области cTFIIB в тройном комплексе окрашен в голубой цвет.Структура cTFIIB в тройном комплексе cTFIIB – TBP – TATA элемент (20) ( Левый ). Структура свободного cTFIIB (23) ( Центр ). Структура N-конца, Zn 2+ связывающая область TFIIB (24) ( справа, ). Атом Zn окрашен в красный цвет. 60 остатков между С-концом Zn 2+ связывающий домен и N-конец cTFIIB являются гибкие и не визуализировались в структурных исследования.
Структуры ТБФ растений (17, 25), дрожжей (18) и человека (19) в комплексе с различными элементами ТАТА (рис.2 А ). Эти три сокристаллические структуры очень похожи и демонстрируют общий механизм индуцированной подгонки (26) белок-ДНК узнавание (G. Patikoglou, J. L. Kim, S.K.B., неопубликованные данные). Связывание ДНК опосредуется изогнутыми антипараллельными β-лист, который обеспечивает большую вогнутую поверхность для малых канавок и Магистральные контакты с элементом ТАТА размером 8 п.н. 5 ‘конец стандарта B-форма ДНК входит в нижнюю часть молекулярного седла, где TBP производит резкий переход к беспрецедентному, частично раскрученному форма правой двойной спирали, индуцированной вставкой двух остатки фенилаланина в первую стадию Т: A основания.После этого расширенная поверхность малой бороздки размотанной, плавно изогнутой ДНК приближается к нижней стороне молекулярного седла, что позволяет прямые взаимодействия между боковыми цепями белка и малой бороздкой края центральной 6 п.н. Второй большой перегиб вызван вставкой двух остатков фенилаланина в основную стадию между двумя последними пары оснований элемента TATA, и имеется соответствующий резкий вернуться к B-форме ДНК. Несмотря на это огромное искажение, Уотсон – Крик спаривание оснований сохраняется повсюду, и, похоже, нет спиральная деформация, индуцированная в ДНК из-за частичного раскручивания. компенсируется правосторонней сверхспирализацией двойной спирали.
Упаковка ДНКв нуклеосомы включает в себя наматывание двойной спирали вокруг октамер гистонов. Последовательно-зависимое позиционирование нуклеосом коррелирует с изгибом A + T-богатых последовательностей к малой бороздке (28, 29), и упаковка элементов ТАТА в нуклеосомы, вероятно, приводит к при сжатии малой канавки, исключающей заедание ТВР. Наоборот, a предварительно сформированный PIC остается транскрипционно активным после нуклеосомы сборки (30), и только рекомбинантный дрожжевой TBP предотвращает репрессия транскрипции, опосредованная нуклеосомами (31).Таким образом сокристаллические структуры комплексов ТВР – ДНК могут обеспечить простой механическое объяснение взаимного исключения упаковки ДНК и транскрипция. Широко распространено мнение, что активаторы транскрипции связаны с промотором проксимальных и / или дистальных энхансерных элементов-мишеней факторы ремоделирования нуклеосом для основных промоторов генов, предназначенных для выражение. После ремоделирования хроматина TFIID сможет распознать элемент TATA и начать сборку PIC (см. 32).
Деформация ДНКпод действием ТВР также может иметь важное значение для координации и / или стабилизация сборки ПОС и взаимодействия активатор-ПОС. Сборка PIC вокруг изгиба может образовывать более компактный комплекс мультипротеин-ДНК. Более того, изгиб ДНК под действием TBP может помочь в образовании петли ДНК, чтобы привести удаленно связанные активаторы транскрипции ближе к коровому промотору для взаимодействия с компонентами ПОС.
Дополнительные биофизические методы были использованы для изучения взаимодействий между TBP и ДНК.Эксперименты по выбору сайта с Acanthamoeba TBP продемонстрировала явное предпочтение сайту очень аналогично изученным кристаллографически (33). Изгиб ДНК с помощью TBP в растворе было подтверждено с помощью анализа круговой перестановки (34). TBP также было показано, что связывание усиливается за счет предварительного изгиба ДНК по направлению к большая канавка и предотвращается предварительным изгибом к малой канавке (35). Кинетика ассоциации ТВР – ДНК изучалась различными методами. (36–38), которые дали результаты, согласующиеся с формированием начального комплекс столкновения с последующей медленной стадией изомеризации с константа скорости второго порядка около 10 6 M -1 · с -1 .После формирования окно TBP – TATA комплекс очень стабилен и измеренный период полужизни дрожжей Время действия комплекса ТБФ – AdMLP в водном растворе составляет примерно 2 ч (36). Наконец, новое исследование химической модификации продемонстрировало, что ядро искажение промотора временно выходит за пределы 3′-конца TATA элемент во время связывания ТВР (39).
Фактор транскрипции IIB
TFIIB — следующий общий фактор инициации для входа в PIC. В образовавшаяся платформа TFIIB-TFIID-ДНК, в свою очередь, распознается комплексом pol II и TFIIF, за которыми следуют TFIIE и TFIIH (рис.1). В vitro исследования с отрицательно свернутым геном иммуноглобулина промотор продемонстрировал, что точное инициирование транскрипции может быть восстановлен с помощью TBP, TFIIB и pol II, что позволяет предположить, что вместе TBP и положение pol II TFIIB (40). Предположительно, энергия, обеспечиваемая отрицательная суперспирализация способствует плавлению промотора на сайт начала транскрипции, которому обычно способствует АТФ-зависимая субъединица ДНК-геликазы TFIIH (см. Ниже). Мутации в TFIIB изменяет стартовые сайты pol II в дрожжах, как и мутации в больших субъединицы pol II, что дает убедительные доказательства ее функции в качестве точный спейсер / мост между TFIID и pol II на промоторе ядра который определяет сайт начала транскрипции (см.20).
Второй этап сборки PIC также оказался поддающимся рентгеновскому облучению. кристаллографическое исследование. Структура элемента TFIIB – TBP – TATA. тройной комплекс был описан в 1995 г. (20) (рис. 2 A ). С-концевой или сердцевинный TFIIB (cTFIIB) представляет собой двухдоменный α-спиральный белок. который является структурным гомологом белка клеточного цикла циклина А (41, 42) (рис.2 B ). Несмотря на эту замечательную конструктивную сходства, нет доказательств того, что TFIIB регулирует активность любая циклин-зависимая киназа.Более того, наличие пара циклин / циклин-зависимая киназа в TFIIH, по-видимому, вызывает вероятность того, что TFIIB будет иметь циклиноподобное поведение, маловероятна.
Тройной комплекс cTFIIB – TBP – ДНК образуется cTFIIB зажатие кислого С-концевого стремени ТВР в его основной щели, и взаимодействуя с основной цепью фосфорибозы до и после центр элемента ТАТА. Первый домен cTFIIB образует нижележащая поверхность тройного комплекса cTFIIB – TBP – ДНК, где вместе с N-концевым доменом TFIIB (24) (проиллюстрировано на рис.2 B ) он мог легко действовать как мост между TBP и pol II, чтобы исправить сайт начала транскрипции. Остальные доступные для растворителя поверхности TBP и TFIIB обширны, обеспечение достаточного количества сайтов узнавания для связывания TAF II s, другие факторы инициации класса II, активаторы транскрипции и соактиваторы. Сама структура комплекса элементов ТВП – ТАТА имеет следующий вид: по существу не изменяется тройным комплексообразованием. cTFIIB распознает предварительно собранный комплекс ТВР-ДНК, включая путь каркас фосфорибозы, созданный беспрецедентной деформацией ДНК индуцируется связыванием ТВР.В дополнение к стабилизации TBP – ДНК комплекса, связывание TFIIB может вносить вклад в полярность элемента ТАТА признание. Если бы TBP связывался с квазисимметричным блоком TATA в неправильная ориентация (т.е. если N-концевая половина молекулярного седла должны были взаимодействовать с 5 ‘концом элемента TATA), основная / гидрофобная поверхность N-концевого хомута сделает неблагоприятные электростатические взаимодействия с основной щелью TFIIB.
Также была определена структура ЯМР раствора одного cTFIIB. (Инжир.2 В ) (23). Хотя каждый домен в ЯМР структура очень похожа на своего аналога в рентгеновской структуре, две структуры демонстрируют разное пространственное расположение два домена. Эти данные предполагают, что олигопептидный линкер между два домена являются гибкими, и этот TFIIB подвергается конформационному изменение при распознавании преформированного комплекса ТВР – ДНК. Таким образом, TFIIB, как и TBP распознает свою цель через индуцированную подгонку (Г. Патикоглу, Дж. Л. Ким, и С.К.Б., неопубликованные данные).
Факторы транскрипции IIE, -IIF и -IIH
После образования комплекса TFIIB – TFIID – ДНК три других общие факторы инициации и pol II завершают растущий PIC. ТФИИФ представляет собой гетеродимер субъединиц массой 30 и 74 кДа (см. обзор в исх. 43). Среди общих инициирующих факторов TFIIF уникален своим способность образовывать очень стабильный комплекс с pol II, называемый pol / F (рис.1). Хотя нет структурного информация, доступная для TFIIF, результаты сайт-ориентированных мутагенез и исследования сшивания белок-ДНК дают некоторые информация о его местонахождении в пределах ПОС.Аланин-сканирование мутагенез ТВР человека выявил единственный остаток, необходимый для TFIIF связка, которая расположена на выпуклой верхней поверхности молекулярного седло на его выходной стороне (44). Фотосшивающие исследования идентифицировали перекрестные связи между субъединицами TFIIF и положениями -5, −15 и −19 (45). Вместе эти данные локализуют TFIIF внутри PIC. к области основного промотора между 3′-концом ТАТА-бокса (позиция -24) и сайт старта транскрипции (рис. 2 А ).TFIIE представляет собой α 2 β 2 гетеротетрамер субъединиц массой 34 и 56 кДа (обзор в исх. 46). Исследования фотоперекрестного сшивания выявили сшивки между Субъединица TFIIE размером 34 кДа и положения -2 и -14 (45), которые локализуют TFIIE к той же части основного промотора, что и TFIIF. TFIIH — это большая мультипротеиновая сборка, состоящая из девяти субъединиц, которые варьируются в масса от 39 до 89 кДа (см. ссылку 47). В отличие от другого генерала факторы инициации, TFIIH поддерживает различную каталитическую активность, включая ДНК-зависимую АТФазу, АТФ-зависимую ДНК-геликазу и серин / треонинкиназа, способная фосфорилировать С-концевой домен большой субъединицы pol II и регулируется субъединица H циклина.По крайней мере, две субъединицы TFIIH (ERCC2 и ERCC3) также являются компонентами механизма эксцизионной репарации ДНК, который предполагает, что мультипротеиновый комплекс TFIIH может также участвовать в Ремонт ДНК (см. Ссылку 48).
Шаги сборки PIC, описанные выше, были установлены в vitro с использованием минимальной системы транскрипции, изображенной на рис. 1 B Верхний . Они не обязательно являются единственным средством из которых можно собрать функциональную ПОС. В последнее время ряд крупных мультипротеиновые комплексы, содержащие pol II и большую часть общих факторы инициации (кроме TFIID и TFIIB) плюс комплекс SRB и другие белки были очищены из ядерного экстракта (см. исх.49). Такие комплексы обычно называют «pol II. голоферменты », что не совсем правильно, потому что они не могут функционировать в одиночку. Эти захватывающие открытия предполагают, что г. vivo PIC можно собрать всего за несколько шагов (например, TFIID плюс ДНК с последующим добавлением TFIIB и затем «pol II холоэнзим », как показано на рис. 1 B ).
Цикл инициации транскрипции РНК Pol II
После завершения сборки PIC и в присутствии нуклеозида трифосфаты, происходит разделение цепи в месте старта транскрипции чтобы дать открытый комплекс, C-концевой домен большой субъединицы pol II фосфорилируется (предположительно киназной субъединицей TFIIH), а pol II инициирует транскрипцию и высвобождается с промотора.Во время удлинения in vitro TFIID может оставаться связанным с основной промотор, поддерживающий повторную инициацию транскрипции с помощью pol II и другие общие факторы инициирования (рис. 1 A ; рассмотрено в исх. 5). Поскольку связывание основного промотора субъединицей ТВР TFIID — это по сути медленный шаг, цикл транскрипции проиллюстрировано на рис. 1 A может представлять механизм инициации pol II in vivo . И потребность в хроматине ремоделирование, которое требует АТФ, и медленная стадия изомеризации во время Деформация ДНК, индуцированная ТВР, будет амортизироваться при многократном инициировании события, если TFIID оставался стабильно связанным с основным промотором между последовательными раундами транскрипции.Этот сценарий особенно привлекателен в контексте сокращенной сборки PIC механизм с участием «холофермента pol II».
Регуляция инициации транскрипции РНК Pol II
Регуляция транскрипции ядерного гена класса II в реакция на сигналы развития или окружающей среды достигается за счет контроль сборки PIC или каталитической эффективности pol II во время инициации, удлинения или прекращения. Когда транскрипционный активаторы взаимодействуют с TAF II с, усиленное рекрутирование и / или наблюдается стабилизация TFIID на промоторе (см. в исх.8). Результаты исследований с гибридными белками, состоящими из TBP, слитый с гетерологичными ДНК-связывающими доменами, предполагает, что TFIID привлечение к промотору может быть этапом, ограничивающим скорость (50–52), который преодолевается взаимодействиями активатор – TAF II . В vivo отпечаток проксимальных областей промотора некоторых специфические для печени гены продемонстрировали, что многие транскрипционные активаторы, по-видимому, связываются одновременно (53), что согласуется с с точки зрения того, что два или более активатора могут оказывать синергетический эффект по транскрипции через согласованное взаимодействие с несколькими компоненты ПОС.Тянь и его коллеги (54, 55) недавно предоставили прямую поддержку этой гипотезе, продемонстрировав, что синергия между двумя разными активаторами (Bicoid и Hunchback) связана к тем же результатам промотора, по крайней мере частично, из определенных взаимодействия с двумя разными Drosophila TAF II s которые увеличивают набор сотрудников TFIID.
В своей простейшей форме белок-белковые взаимодействия, регулирующие pol II активность вовлекает компоненты преинициативного комплекса (TBP, TAF II s, TFIIB, pol II, TFIIF, TFIIE и TFIIH) и активаторы транскрипции (связанные либо с проксимальным, либо с дистальным промотором). энхансерные элементы).Наша текущая картина активатора – TFIID взаимодействий предполагает, что TAF II s можно рассматривать как большой мультипротеиновый комплекс, который находится поверх TBP и интегрирует сигналы от многих активаторов и коактиваторов, не относящихся к TAF II . В оставшиеся общие факторы инициации и pol II представляют разные мишени в PIC для взаимодействия с транскрипционными активаторы. Действительно, кажется вероятным, что каждый компонент ПОС мишень хотя бы одного активатора транскрипции во время транскрипция одного или нескольких из примерно 100000 классов II промоторы ядерных генов.Косвенное взаимодействие между PIC и активаторы транскрипции, опосредованные не-TAF II соактиваторы также наблюдались (рис. 1 B ). Коактиваторы, такие как человеческий PC4, человеческий OCA-B и дрожжевой SRB комплекс, может служить переходником между активаторами и базальными факторами (рассмотрено в ссылке 8).
Фактор транскрипции IIA
TFIIA был впервые описан как общий фактор инициации (7) и изначально считалось необходимым для транскрипции многих, если не все промоторы ядерных генов класса II.После обширных механистическая характеристика и клонирование генов, кодирующих субъединицы TFIIA, однако теперь ясно, что TFIIA лучше всего определяется как коактиватор, поддерживающий регуляцию транскрипции pol II (рассмотрено в ссылке 3). На ранней стадии сборки PIC TFIIA может связываться с и стабилизировать комплексы TFIID – ДНК или TFIIB – TFIID – ДНК, позволяя им предотвращать пагубные эффекты подавляющего отрицательные кофакторы и усиливают стимулирующее действие активаторы транскрипции (см.56).
Недавно была обнаружена структура тройного комплекса элементов TFIIA – TBP – TATA. определено методом рентгеновской кристаллографии (21, 22) (рис. 2 А ). TFIIA дрожжей состоит из двух α / β субъединицы 14 и 32 кДа, которые образуют интимный гетеродимер через 12-ниточная β-цилиндрическая структура. Тройной комплекс образован TFIIA распознает N-концевую скобу TBP и взаимодействует с остов фосфорибозы перед элементом ТАТА на противоположной стороне грань двойной спирали cTFIIB (рис.2 А ). В качестве в комплексе cTFIIB – TBP – ДНК TFIIA распознает предварительно сформированные Комплекс TBP – ДНК, объясняющий стабилизацию комплекса TFIID – ДНК с помощью TFIIA.
Когда структуры cTFIIB – TBP – ДНК и TFIIA – TBP – ДНК комплексы объединяются для создания модели TFIIA – TFIIB – TBP – ДНК четвертичный комплекс (рис. 3), механизм по которые TFIIB и TFIIA действуют синергетически в стабилизации TFIID-ДНК комплекс можно рационализировать. Вместо того, чтобы взаимодействовать друг с другом непосредственно основные поверхности TFIIB и TFIIA контактируют с отрицательно заряженный остов фосфорибозы на противоположных сторонах двойная спираль непосредственно перед элементом ТАТА.Модель Комплекс TFIIA – TFIIB – TBP – ДНК также дает критическую информацию о роль TFIIA как коактиватора или моста между транскрипционными активаторы и ПОС. Обе субъединицы TFIIA образуют вышестоящий поверхность тройного комплекса TFIIA – TBP – ДНК, где они находятся доступны для взаимодействия с активаторами транскрипции, связанными с промоторные проксимальные или дистальные энхансерные элементы. Следовательно, это не удивительно, что остатки на поверхности ТВР, которые участвуют в контакты с TFIIA необходимы для активации транскрипции в vivo (56, 57).
Модель комплекса TFIIA – TFIIB – TBP – ДНК на основе на структуры элемента cTFIIB – TBP – TATA (20), а Комплексы TFIIA – TBP – TATA элемент (21, 22) (см. Рис. 2 А ). Сайт начала транскрипции помечен +1. Схема цветового кодирования такая же, как на рис. 2 A . ( Верхний ) Если смотреть вдоль оси TBP приблизительного внутримолекулярная симметрия сверху седла. ( Нижний ) Вид снизу на молекулярное седло.
Выводы и перспективы
Прошло более четверти века с тех пор, как сложность транскрипции эукариот впервые было обнаружено открытием Редера трех РНК-полимераз (58).С тех пор технически сложно биохимическая работа и элегантные генетические исследования определили и функционально охарактеризованы многие компоненты, которые вместе облегчают и регулируют производство Pol II матричной РНК. Трехмерные структуры ТВП и его комплекса с ядром промотор, cTFIIB, TFIIBn и TFIIB – TBP – ДНК и TFIIA – TBP – ДНК тройные комплексы выявили новые взаимодействия белок-ДНК, и детальная механистическая оценка того, как эти полипептиды поддерживают инициация транскрипции.Структурные биологи сейчас занимаются даже более крупные сборки факторов транскрипции, и есть все основания для полагаем, что скоро мы увидим структуры TFIIE, TFIIF, TFIID, TFIIH, и РНК pol II. Биологи факторов транскрипции в настоящее время руководят их усилия по решению проблемы понимания того, как транскрипция инициация контролируется на уровне отдельного гена. Там есть значительные доказательства того, что PIC и активаторы транскрипции и коактиваторы могут собираться на промоторе в стереоспецифический нуклеопротеидный комплекс или «транскриптосома», поддерживающий активация транскрипции (см.59).
Другой важной проблемой, которую необходимо решить, является необходимость понять сложное взаимодействие между упаковкой ДНК и транскрипция. Неожиданно недавние кристаллографические исследования задокументированные прямые структурные связи между факторами транскрипции и гистоновые белки (рис. 4), предполагая, что макромолекулярные машины, отвечающие за упаковку ДНК и транскрипция на каком-то уровне эволюционно связана. В структурные взаимосвязи, показанные на рис.4 также вызывают интригующие вопросы, касающиеся механизмов, с помощью которых гистоноподобная транскрипция факторы работают (см. ссылку 64). TFIID может содержать TAF II субструктура, которая напоминает октамер гистонов и опосредует некоторые неспецифические взаимодействия TFIID с ДНК (11). Прямые доказательства обертывания ДНК вокруг TFIID были получены Roeder и соавторы (65), которые продемонстрировали TAF II -DNA сшивает непосредственно перед элементом ТАТА и ниже по потоку элемент ТАТА, простирающийся в 5′-нетранслируемую область гена, и TFIID-индуцированная суперспирализация ДНК замкнутой кольцевой плазмиды.В напротив, связывание ТВР с той же плазмидой не изменяет связывание число, потому что суперспирализация ДНК TBP компенсируется частичной раскручивание двойной спирали (см. ссылку 25). Наконец, структурное сходство ядерного фактора гепатоцитов (HNF) -3γ и гистон H5 может иметь функциональное значение. Связывание HNF-3 с двумя соседние высокоаффинные сайты в энхансере гена сывороточного альбумина мыши (66), как было показано, индуцируют фазирование расположения нуклеосом. внутри энхансера (27).
Структурное сходство между транскрипцией факторы и гистоновые белки. ( Верхний ) Гетеротетрамерный сборка N-концевых частей двух Drosophila TAF II с (dTAF II 42 / dTAF II 62) 2 (60) и соответствующий вид гетеротетрамера гистона h4 / h5, производного из структуры октамера гистонов (61) (дополнительная N-концевая спираль h4, визуализированная в этом исследовании, была опущена для ясность).( Нижний ) ДНК-связывающий домен гепатоцита ядерный фактор-3γ (62), и соответствующий вид глобулярной домен линкерного гистона H5 (GH5) (63).
Благодарности
Мы благодарим докторов наук. Г. Аренц, С. Бэгби, Дж. Гейгер, М. Икура, Дж. Л. Ким, Э. Н. Мудрианакису, П. Б. Сиглеру, М. Саммерсу и X. Се за помощь с фигурная подготовка. Эта работа была поддержана Ховардом Хьюзом. Медицинский институт (S.K.B.) и выпускник Университета Рокфеллера Товарищество (Д.Б.Н.).
Сноски
↵ Текущий адрес: Клеточная биохимия и биофизика Программа, Мемориальный онкологический центр Слоуна – Кеттеринга, Нью-Йорк, штат Нью-Йорк 10021.
↵ Кому обращаться с запросами на перепечатку: Университет Рокфеллера, 1230 York Avenue, New York, NY 10021. Электронная почта: Берли rockvax.rockefeller.edu.
Сокращения: pol II, полимераза II; PIC, преинициация сложный; AdMLP, главный поздний промотор аденовируса; cTFIIB, терминал C или ядро TFIIB.
- Авторские права © 1997, Национальная академия наук США
Что такое экспрессия генов? | Факты
Экспрессия гена — это процесс, с помощью которого инструкции в нашей ДНК преобразуются в функциональный продукт, такой как белок.
- Когда информация, хранящаяся в нашей ДНК, преобразуется в инструкции по созданию белков или других молекул, это называется экспрессией генов.
- Экспрессия гена — это строго регулируемый процесс, который позволяет клетке реагировать на изменение окружающей среды.
- Он действует как переключатель включения / выключения для управления производством белков, а также как регулятор объема, который увеличивает или уменьшает количество производимых белков.
- Есть два ключевых этапа создания белка: транскрипция и трансляция.
Транскрипция
- Транскрипция — это когда ДНК в гене копируется для получения транскрипта РНК, называемого информационной РНК (мРНК).
- Это осуществляется ферментом, называемым РНК-полимеразой, который использует доступные основания из ядра клетки для формирования мРНК.
- РНК — это химическое вещество, сходное по структуре и свойствам с ДНК, но оно имеет только одну цепь оснований, а вместо основания тимина (T) РНК имеет основание, называемое урацилом (U).
Иллюстрация, показывающая процесс транскрипции.
Изображение предоставлено: Genome Research Limited
Трансляция
- Трансляция происходит после того, как информационная РНК (мРНК) передала транскрибированное «сообщение» от ДНК к фабрикам по производству белка в клетке, называемым рибосомами.
- Сообщение, передаваемое мРНК, считывается молекулой-носителем, называемой транспортной РНК (тРНК).
- МРНК считывается по трем буквам (кодону) за раз.
- Каждый кодон определяет конкретную аминокислоту. Например, три основания «GGU» кодируют аминокислоту под названием глицин.
- Поскольку существует только 20 аминокислот, но 64 потенциальных комбинации кодонов, более одного кодона могут кодировать одну и ту же аминокислоту. Например, кодоны «GGU» и «GGC» кодируют глицин.
- Каждая аминокислота специфически прикрепляется к своей собственной молекуле тРНК.
- Когда последовательность мРНК считывается, каждая молекула тРНК доставляет свою аминокислоту на рибосому и временно связывается с соответствующим кодоном на молекуле мРНК.
- После связывания тРНК высвобождает свою аминокислоту, и все соседние аминокислоты объединяются в длинную цепь, называемую полипептидом.
- Этот процесс продолжается до образования белка.
- Белки выполняют большинство активных функций клетки.
Иллюстрация, показывающая процесс перевода.
Изображение предоставлено: Genome Research Limited
Эта страница последний раз обновлялась 21.07.2021
Всесторонняя транскрипция генома человека дает тысячи ранее не идентифицированных длинных межгенных некодирующих РНК
Abstract
Известные экзоны гена, кодирующего белок, составляют менее 3% генома человека. Остальные 97% — это в основном неизведанная территория, охарактеризована лишь небольшая часть.Недавнее наблюдение транскрипции на этой межгенной территории стимулировало дебаты о степени межгенной транскрипции и функциональности этих межгенных РНК. Здесь мы непосредственно наблюдали с большим набором данных RNA-seq, охватывающих широкий спектр типов тканей человека, что большая часть генома действительно транскрибируется, подтверждая недавние наблюдения проекта ENCODE. Кроме того, используя сборку транскриптома de novo из этих данных РНК-seq, мы обнаружили, что межгенные области кодируют гораздо более длинные межгенные некодирующие РНК (lincRNAs), чем описано ранее, что помогает устранить несоответствие между огромным количеством наблюдаемой межгенной транскрипции и ограниченное количество ранее известных линкРНК.В общей сложности мы идентифицировали десятки тысяч предполагаемых lincRNAs, экспрессируемых как минимум в одной копии на клетку, что значительно расширило предыдущие наборы аннотаций lincRNA. Эти lincRNA специфически регулируются и сохраняются, а не являются продуктом транскрипционного шума. Кроме того, lincRNA сильно обогащены SNP, ассоциированными с признаками, что указывает на новый механизм, с помощью которого могут функционировать области, ассоциированные с межгенными признаками. Эти результаты позволят обнаруживать и исследовать новые межгенные функциональные элементы.
Сведения об авторе
Большая часть генома человека состоит из межгенных последовательностей, областей между генами. Когда-то считалось, что межгенная последовательность представляет собой «мусорную ДНК», не имеющую отношения к транскрипции, но недавно стало очевидно, что межгенные области могут транскрибироваться. Однако объем, природа и идентичность этой межгенной транскрипции остаются неизвестными. Здесь, анализируя большой набор данных RNA-seq, мы обнаружили, что транскрибируется> 85% генома, что позволяет нам создать полный каталог важного класса межгенных транскриптов: длинных межгенных некодирующих РНК (lincRNA).Мы обнаружили, что геном кодирует гораздо больше lincRNA, чем было известно ранее. Ключевым вопросом в этой области является то, являются ли эти межгенные транскрипты функциональным или транскрипционным шумом. Мы обнаружили, что идентифицированные нами lincRNA обладают многими характеристиками, несовместимыми с шумом, включая специфическую регуляцию их экспрессии, наличие консервативной последовательности и доказательства регулируемого процессинга. Кроме того, эти линкРНК сильно обогащены межгенными последовательностями, которые, как ранее было известно, функциональны при человеческих свойствах и заболеваниях.Это исследование обеспечивает важную основу, с помощью которой можно идентифицировать и охарактеризовать функциональные элементы в межгенных регионах, облегчая будущие усилия по пониманию роли межгенной транскрипции в здоровье и болезнях человека.
Образец цитирования: Hangauer MJ, Vaughn IW, McManus MT (2013) Всесторонняя транскрипция генома человека производит тысячи ранее не идентифицированных длинных межгенных некодирующих РНК. PLoS Genet 9 (6): e1003569. https: // doi.org / 10.1371 / journal.pgen.1003569
Редактор: Джон Л. Ринн, Институт Броуда Массачусетского технологического института и Гарвард, Соединенные Штаты Америки
Поступила: 28 сентября 2012 г .; Дата принятия: 1 мая 2013 г .; Опубликован: 20 июня 2013 г.
Авторские права: © 2013 Hangauer et al. Это статья в открытом доступе, распространяемая в соответствии с условиями лицензии Creative Commons Attribution License, которая разрешает неограниченное использование, распространение и воспроизведение на любом носителе при условии указания автора и источника.
Финансирование: Эта работа финансировалась грантом NIH 5U01ES017154 как часть NIH Human Epigenome Atlas UCSF-UBC Reference Epigenome Mapping Center (MTM), грант NIH U01CA168370 как часть NIH Bay Area Cancer Target Discovery and Development Network ( MTM), PBBR New Frontier Research Award (MTM) и Сьюзан Г. Комен за стипендию для получения докторской степени KG1101214 (MJH). Финансирующие организации не играли никакой роли в дизайне исследования, сборе и анализе данных, принятии решения о публикации или подготовке рукописи.
Конкурирующие интересы: Авторы заявили об отсутствии конкурирующих интересов.
Введение
Большая часть генома человека состоит из межгенных последовательностей. Когда-то упоминавшаяся как «мусорная ДНК», теперь ясно, что функциональные элементы существуют в межгенных областях. Фактически, полногеномные ассоциативные исследования показали, что примерно половина всех геномных областей, ассоциированных с заболеваниями и признаками, являются межгенными [1]. Хотя некоторые из этих областей могут функционировать исключительно как элементы ДНК, теперь известно, что межгенные области могут транскрибироваться [2] — [7], и появился постоянно растущий список функциональных некодирующих генов РНК внутри межгенных областей [8].
Несмотря на этот прогресс, полное понимание степени межгенной транскрипции и идентичности этих транскриптов остается неуловимым. Первые попытки проанализировать степень и природу межгенной транскрипции использовали технологию тайлинг-массива [2] — [5]. Эти исследования подтвердили, что межгенная транскрипция является повсеместной, но озабоченность по поводу кросс-гибридизации вызвала дискуссию по поводу данных [9] — [12]. Кроме того, чтобы избежать технических трудностей, связанных с анализом повторяющейся последовательности с использованием тайлинговых массивов, исследования были ограничены оценкой менее половины генома.Совсем недавно несколько исследований были сосредоточены на оценке степени межгенной транскрипции с использованием подходов, основанных на секвенировании, но за исключением недавно опубликованных результатов проекта ENCODE [13], [14], эти исследования до сих пор ограничивались очень узкими предварительно отобранные участки генома и небольшое количество тканей [6], [7]. Преодолевая эти предыдущие недостатки, проект ENCODE использовал анализ последовательности РНК в сочетании с другими технологиями для профилирования 15 линий клеток человека, обеспечивая доказательства транскрипции в 83.7% генома человека и твердо устанавливающие реальность всепроникающей транскрипции [14].
Длинные межгенные некодирующие РНК (lincRNA) определяются как межгенные (относительно текущих аннотаций генов) транскрипты длиной более 200 нуклеотидов, которые не обладают способностью кодировать белок. LincRNAs, как известно, выполняют множество функций посредством различных механизмов, начиная от регуляции эпигенетических модификаций и экспрессии генов до действия в качестве каркаса для белковых сигнальных комплексов [8], [15].Первые попытки генерировать наборы аннотаций lincRNA либо профилировали lincRNAs, специфичные для небольшого количества тканей, либо требовали, чтобы транскрипты обладали специфическими структурными особенностями, такими как сплайсинг и полиаденилирование [16] — [18]. Консорциум GENCODE (GENCODE v7) вручную курировал около пяти тысяч lincRNA, которые не ограничены конкретными тканями или структурными особенностями, однако этот набор аннотаций содержит только небольшую часть всех lincRNA, поскольку он не использует данные RNA-seq для идентификации новые записи [19], [20].Ограниченный масштаб текущих аннотаций lincRNA, включая GENCODE, явно несовместим с огромным количеством межгенной транскрипции, наблюдаемой в проекте ENCODE. Следовательно, следует ожидать, что геном кодирует гораздо больше lincRNA, чем известно в настоящее время.
Чтобы преодолеть разрыв между наблюдением всеобъемлющей межгенной транскрипции в рамках проекта ENCODE и ограниченным в настоящее время набором аннотированных lincRNAs, мы провели анализ уникального набора данных RNA-seq, полученных как из новых, так и из опубликованных наборов данных, которые дополняют и значительно расширяет предыдущие исследования [14], [16], [19].Этот анализ привел к четкому подтверждению наблюдений всеобъемлющей транскрипции в геноме человека в рамках проекта ENCODE [14]. Более того, анализ ранее аннотированных предполагаемых lincRNAs, включая таковые из проекта ENCODE [19], в дополнение к открытию de novo новых lincRNAs из данных RNA-seq привел к составлению наиболее полного каталога человеческих lincRNAs. Благодаря расширению выборки тканей и ослаблению ограничений на структуру транскриптов, мы находим значительно больше lincRNAs, чем все предыдущие наборы аннотаций lincRNA вместе взятые.Наш анализ показал, что эти lincRNAs демонстрируют многие особенности, согласующиеся с функциональностью, в отличие от предшествующих заявлений о том, что межгенная транскрипция является в первую очередь продуктом транскрипционного шума [12]. В целом, наши результаты подтверждают недавние сообщения о всеобъемлющей транскрипции в геноме человека и демонстрируют, что межгенная транскрипция приводит к продукции большого количества ранее неизвестных lincRNAs. Мы предоставляем этот значительно расширенный набор аннотаций lincRNA в качестве важного ресурса для изучения межгенных функциональных элементов в здоровье и болезнях человека.
Результаты
Количественное определение степени транскрипции генома человека
Мы проанализировали шесть новых наборов данных RNA-seq, созданных в рамках Атласа эпигенома человека (http://www.genboree.org/epigenomeatlas/index.rhtml), и 121 ранее опубликованный набор данных RNA-seq, представляющих 23 ткани человека в различных условиях. и состоит из более чем 4,5 миллиардов однозначно отображаемых операций чтения (таблица S1). Этот набор данных RNA-seq позволил выявить как редкие, так и тканеспецифичные события транскрипции, которые в противном случае не могли бы быть обнаружены.В отличие от ограниченного охвата предыдущих исследований мозаичных массивов [2] — [5], мы проанализировали гораздо большую часть (83,4%) генома, с которой считывания RNA-seq могут быть однозначно сопоставлены, что обеспечивает более широкое представление о транскриптоме. . При пороге одного чтения РНК-секвенции мы наблюдали картирование чтения на 78,9% генома, и, если принимать во внимание дополнительные доказательства транскрипции, включая полные структуры известных генов, сплайсированных EST и кДНК, мы обнаружили доказательства того, что 85,2% генома транскрибируется (рис. 1А).Этот результат полностью согласуется с недавно опубликованными результатами проекта ENCODE, в котором были обнаружены доказательства транскрипции 83,7% генома [14]. Интересно, что даже с 4,5 миллиардами отображенных считываний мы наблюдаем увеличение геномного покрытия при каждом более низком пороге считывания, что означает, что даже большая глубина считывания может выявить еще более высокий геномный охват. (Рисунок S1).
Рисунок 1. Человеческий межгенный транскриптом.
(A) 85,2% генома имеет свидетельства транскрипции, при этом RNA-seq reads отображается непосредственно на 78.9% геномной последовательности. Остающийся геномный охват состоит из известных генов, сплайсированных EST и сплайсированных кДНК. Серый кружок представляет собой часть генома (83,4%), которая однозначно картируется с помощью считываний RNA-seq. (B) Распределение уровней экспрессии экзона, интрона и межгенной области, кодирующего белок (ген NM). В регионах с высоким уровнем выраженности большая часть базовых вызовов появляется на более высоких глубинах чтения. Экзоны генов, кодирующих белок, имеют наибольшую долю оснований с высокой глубиной считывания, в то время как интроны и межгенные области имеют относительно больше оснований с низкой глубиной считывания, хотя каждый из них содержит много высоко экспрессируемых участков.Базовые вызовы = (# геномных позиций на определенной глубине чтения) (глубина чтения). (C) Большая часть межгенной транскрипции происходит за пределами аннотированных генов некодирующей РНК. Сравнивается доля вызовов межгенных оснований в генах некодирующей РНК RefSeq (NR-гены) по сравнению с другими межгенными участками. В (A – C) рассматриваются только однозначно отображаемые части генома (см. Методы).
https://doi.org/10.1371/journal.pgen.1003569.g001
Как и ожидалось, экзоны генов, кодирующих белок, содержат наибольшую долю высокоэкспрессируемых оснований (рис. 1B), а также непропорционально большую долю от общего числа считываний по сравнению с их небольшое (<3%) количество геномной последовательности (Рисунок S2).Однако многие регионы с высокой экспрессией действительно существуют внутри межгенных регионов, гораздо больше, чем это объясняется текущими аннотациями генов некодирующей РНК (Figure 1C). Мы рассудили, что эта неучтенная межгенная транскрипция должна происходить из новых межгенных транскриптов, и затем направили наши усилия на идентификацию и анализ этих транскриптов.
Открытие большого количества новых LincRNA
Мы предположили, что большая часть межгенной транскрипции, не учитываемая ранее аннотированными транскриптами, происходит от новых lincRNAs.Мы пришли к выводу, что, поскольку экспрессия lincRNA, как известно, очень тканеспецифична [16], широта тканей и условий, отобранных в анализируемых здесь наборах данных RNA-seq, может помочь в обнаружении lincRNA.
Мы использовали этот большой набор данных RNA-seq в сочетании с предыдущими наборами аннотаций некодирующих РНК для создания наиболее полного каталога lincRNAs (рисунок 2A). Чтобы создать этот каталог lincRNA, мы сначала собрали известные и предполагаемые аннотированные lincRNA. Мы собрали некодирующие РНК, присутствующие в общедоступных базах данных, включая GENCODE v6, и из литературных источников [16], [18], в результате чего был получен набор из 351 940 транскриптов.Кроме того, мы выполнили сборку транскриптома de novo на каждом из наборов данных RNA-seq (Таблица S2) для генерации 6 833 809 собранных транскриптов de novo . Как ранее аннотированные, так и собранные de novo транскриптов были отфильтрованы для удаления транскриптов, перекрывающих гены, кодирующие белок, известные гены РНК, не кодирующие lincRNA, и псевдогены. Транскрипты длиной более 200 нуклеотидов дополнительно фильтровали для удаления любых транскриптов, содержащих (или перекрывающих любой другой транскрипт, содержащий) открытую рамку считывания (ORF) длиной более 100 аминокислот.Из-за опасений, что около собранных de novo транскриптов могут быть неаннотированными расширениями соседних генов, кодирующих белок, как это недавно наблюдалось для части длинных некодирующих РНК GENCODE [19], мы создали дополнительный фильтр для удаления транскриптов, связанных с соседними генами посредством РНК-seq читает. Для этого мы расширили аннотации ссылок на гены, кодирующие белок, используя сборку транскриптомов de novo и удалили транскрипты, перекрывающие эти расширенные генные структуры (см. Методы, набор данных S1).
Рисунок 2. Открытие линкРНК.
(A) Открытие lincRNA включало сборку de novo транскриптов из данных RNA-seq и компиляцию аннотированных и предполагаемых некодирующих РНК (см. Методы), за которыми следовала серия фильтров, предназначенных для удаления всех известных и новых кодирующих белков. транскрипты и некодирующие РНК, отличные от lincRNA. Только межгенные некодирующие транскрипты длиной не менее 200 нуклеотидов и экспрессированные, по крайней мере, по одной копии на клетку, в конечном итоге были аннотированы как lincRNAs.(B) Анализ данных рибосомного профилирования показывает, что каталог lincRNA состоит из некодирующих транскриптов. Максимальное соотношение окна 30 п.н. рибосомы HeLa / считываний РНК-seq [22] нанесено на график для экзонов lincRNA, 3 ‘UTR и кодирующих последовательностей (CDS). * P <2.2E-16; усы увеличивают межквартильный размах в +/- 1,5 раза, а точки представляют собой выбросы. (C) Компьютерный анализ способности lincRNA кодировать белок показывает отсутствие способности кодировать белок. На графике нанесено кумулятивное распределение оценок PhyloCSF [40] для lincRNA и RefSeq NM генов.Более высокие оценки соответствуют более высокой прогнозируемой способности кодирования.
https://doi.org/10.1371/journal.pgen.1003569.g002
На последнем этапе мы удалили транскрипты, выраженные с количеством фрагментов на килобазу транскрипта на миллион отображенных считываний (FPKM) <1, порог примерно эквивалентен одному копировать на ячейку [21] (Таблица S1). Для уменьшения избыточности и с целью идентификации «генов» линкРНК, а не потенциально избыточных перекрывающихся «транскриптов», оставшиеся транскрипты были объединены, если они имели хотя бы один общий экзон (см. Методы), в результате получилось 53 864 различных предполагаемых lincRNA с FPKM> 1, 3676 линкРНК при FPKM> 10 и 925 линкРНК при FPKM> 30 (набор данных S2 и рисунок S3).Удивительно, но более 94% окончательного набора объединенных lincRNA на каждом уровне экспрессии состоит исключительно из новых собранных de novo транскриптов, обнаруженных на основе данных RNA-seq в этом исследовании (таблица S3 и набор данных S2). Эти lincRNAs распределены по всей межгенной последовательности вместо того, чтобы кластеризоваться рядом с аннотированными в настоящее время генами. 58,1% FPKM> 1 lincRNA, 61,9% FPKM> 10 lincRNA и 67,7% FPKM> 30 lincRNA находятся на расстоянии более 30 тысяч оснований от ближайшего гена, кодирующего белок, на любой цепи.Мы аннотировали lincRNAs как принадлежащие к одной и той же «группе» (см. Методы), если они находятся в пределах 1 килобаз друг от друга, чтобы учесть возможность того, что некоторые проксимальные аннотации lincRNA могут быть частичными структурами более крупных транскриптов (см. Обсуждение). Это группирование привело к 35 585 отдельным группам lincRNA при FPKM> 1, 2970 при FPKM> 10 и 764 при FPKM> 30, и lincRNA в каталоге названы в соответствии с этими группами (набор данных S2). Эти аннотации, вероятно, будут неполными из-за ограничений сборки транскриптов из данных RNA-seq; действительно, некоторые аннотации могут быть фрагментами более крупных перекрывающихся транскриптов lincRNA.Следовательно, фактическое количество независимых lincRNAs может отличаться от приведенных выше чисел, и в будущем необходима работа для более полного определения полных, независимых аннотаций транскриптов lincRNA (см. Обсуждение).
Оценка подхода к фильтрации LincRNA
Мы оценили строгость, с которой наш процесс фильтрации удалял кодирующие белки транскрипты, анализируя данные рибосомного профилирования клеток HeLa (рис. 2B) [22]. Как и ожидалось, lincRNAs напоминают экзоны 3′-нетранслируемой области генов, кодирующих белок, с очень небольшим количеством транскриптов, демонстрирующих значительное взаимодействие с рибосомой.Это открытие согласуется с недавним наблюдением, что длинные некодирующие РНК GENCODE (подмножество нашего каталога) обычно лишены основанных на масс-спектрометрии доказательств трансляции [23]. Напротив, недавнее исследование показало, что многие ранее аннотированные lincRNA мыши связывают рибосомы [24]. Хотя биологическое значение этого несоответствия неизвестно, оно может быть результатом различий в строгости подхода к фильтрации, используемого при генерации рассматриваемых аннотаций lincRNA.Дальнейшее подтверждение строгости наших фильтров, вычислительный анализ потенциала кодирования белка с использованием программы PhyloCSF показал, что наш набор отфильтрованных lincRNAs не имеет предсказанной способности кодирования белка (рис. 2C). Из этих анализов мы пришли к выводу, что наш подход к фильтрации эффективно удалил кодирующие белки транскрипты из каталога.
Дополнительные каталоги и ресурсы LincRNA
В то время как оставшаяся часть этого исследования посвящена этому каталогу предполагаемых lincRNAs (Dataset S2), мы предоставили несколько альтернативных каталогов lincRNA.Они включают объединенный каталог lincRNAs, идентифицированных в этом исследовании, объединенных (см. Методы) с набором дополнительных lincRNAs, идентифицированных в Cabili, et al. [16], которые прошли все наши фильтры, за исключением того, что они не экспрессировались при FPKM> 1 ни в одном из проанализированных здесь наборов данных RNA-seq. Добавленные lincRNA экспрессируются при FPKM> 1 в одном или нескольких наборах данных RNA-seq, проанализированных в Cabili et al. [16], которые полностью отличаются от анализируемых здесь наборов данных и, следовательно, могут быть подлинными lincRNAs по нашим критериям.Этот каталог (Dataset S3) включает 54 784 lincRNA с FPKM> 1 (920 дополнительных lincRNA по сравнению с Dataset S2), 3764 lincRNA с FPKM> 10 (88 дополнительных lincRNA) и 942 lincRNA с FPKM> 30 (17 дополнительных lincRNA). Кроме того, мы включили каталог сплайсированных lincRNA, которые экспрессируются при FPKM> 1, по крайней мере, в одном наборе данных (4576 lincRNAs, Dataset S4), из которых 61% состоит исключительно из собранных de novo транскриптов, обнаруженных в этом исследовании. Мы также составили каталог lincRNAs, экспрессируемых при FPKM> 1, по крайней мере, в двух наборах данных (26 455 lincRNAs, Dataset S5), из которых 97% представляют собой исключительно собранных de novo транскриптов, обнаруженных здесь.Кроме того, был включен альтернативный каталог lincRNA, содержащий только те lincRNA, экспрессируемые значительно выше, чем случайно выбранные межгенные области (см. Методы) (5267 lincRNA, наборы данных S6, S7). Кроме того, в качестве дополнительного ресурса мы предоставляем уровень экспрессии (количество считываний FPKM и raw RNA-seq) всех lincRNA (в наборе данных S2) и генов, кодирующих белок RefSeq, во всех 127 наборах данных RNA-seq (набор данных S8).
LincRNAs специально регулируются
Степень функциональности межгенной транскрипции остается неопределенной и спорной [9] — [12], [25].Чтобы оценить, регулируются ли lincRNAs, идентифицированные в настоящем исследовании, специфически, в отличие от транскрипционного шума, мы определили, несут ли гены lincRNA канонические эпигенетические метки для активации и репрессии, мотивируя это тем, что шумовые транскрипты не должны иметь когерентных паттернов эпигенетической модификации. В соответствии с наблюдениями, основанными на более ранних аннотациях длинных некодирующих РНК [18], [19], [26], [27], анализ данных ChIP-seq и RNA-seq [28], [29] показал, что каталог lincRNAs показывает паттерны эпигенетической модификации аналогичны генам, кодирующим белок (рис. 3А).Активирующие гистоновые метки, h4K4me3 и h4K36me3, оба значительно обогащены высокоэкспрессируемыми lincRNA. Точно так же репрессивная метка h4K27me3 значительно обогащена низкоэкспрессируемыми lincRNA. Таким образом, экспрессия lincRNA, по-видимому, специфически регулируется.
Рис. 3. LincRNA обладают характеристиками, несовместимыми с шумом транскрипции.
(A) Данные ChIP-seq и RNA-seq из клеток IMR90 [28], [29] были проанализированы на предмет lincRNAs и RefSeq NM генов. * P = 4.01E-7, ** P = 4,52E-9, *** P = 2,43E-14, **** P <2,2E-16; P = 0,137 для линкРНК h4K9me3; усы простираются до +/- 1,5-кратного межквартильного размаха или до самой экстремальной точки данных. (B) Сравнивались значения FPKM LincRNA в полиА + -специфичных и полиА- специфичных библиотеках РНК-seq в ESCs H9 и клетках HeLa [46]. Транскрипты с RNA-seq считываниями во всех четырех наборах данных и с FPKM> 1 по крайней мере в одной из двух фракций для каждого типа клеток были проанализированы (16 819 генов NM и 127 lincRNA).На графике нанесены индивидуальные соотношения генов lincRNA и NM FPKM во фракциях полиА + / полиА-. Значение корреляции Пирсона для lincRNAs = 0,622 ( P = 5,551E-15) и для генов NM = 0,702 ( P <2,2E-16). (C) Были определены максимально консервативные окна размером 50 п.н. в каждом гене NM, lincRNA и повторяющемся элементе (неконсервативные контрольные последовательности). Для сравнения указаны максимально консервативные окна размером 50 п.н. 12 функциональных lincRNA человека.
https: // doi.org / 10.1371 / journal.pgen.1003569.g003
Если lincRNAs специфически регулируются на уровне транскрипции, ожидается, что уровни их экспрессии специфичны для их тканевого источника. Действительно, предыдущие исследования lincRNAs показали, что lincRNAs обнаруживают очень сильную тканеспецифичную экспрессию [16], [19]. Чтобы проверить, остается ли это верным с нашим расширенным набором lincRNA, мы выполнили неконтролируемую иерархическую кластеризацию, используя уровни экспрессии lincRNA в реплицируемых наборах данных RNA-seq из различных тканей (рисунок S4).Реплики каждого типа ткани сильно сгруппированы вместе, что указывает на то, что дифференциальная экспрессия lincRNA действительно воспроизводимо тканеспецифична, поддерживая специфическую регуляцию экспрессии lincRNA.
LincRNAs не нуждаются в полиаденилировании, чтобы быть функциональными [30]. По этой причине мы включили в наш анализ многие библиотеки RNA-seq, которые не были отобраны по полиА +. Фактически, более ранние исследования тайлингового массива показали, что межгенные транскрипты имеют тенденцию быть биморфными; то есть они появляются как во фракциях полиА +, так и полиА-, в отличие от транскриптов, кодирующих белок, которые в основном являются полиА + [3].Недавно опубликованные результаты ENCODE подтверждают этот вывод [14], [19]. В соответствии с этими исследованиями мы обнаружили, что статус полиаденилирования lincRNAs в нашем каталоге воспроизводимо биморфен для нескольких типов клеток, в то время как кодирующие белок транскрипты сильно обогащены в образце polyA +. Воспроизводимость этого биморфного состояния lincRNA предполагает, что полиаденилирование lincRNA регулируется и что многие lincRNA существуют, по крайней мере, частично в виде неполиаденилированных транскриптов (Рисунок 3B и Рисунок S5).Это открытие указывает на то, что будущие исследования lincRNAs не должны игнорировать фракцию неполиаденилированной РНК.
Затем мы оценили, консервативны ли lincRNA. Было замечено, что lincRNAs могут содержать консервативные мотивы, связанные вместе неконсервативной последовательностью [25], [31], [32]. Таким образом, мы оценили сохранение lincRNA, используя окно сканирования 50 п.н. (Рисунок 3C, Рисунок S6 и Таблица S4). В соответствии с предыдущими исследованиями lincRNAs обнаруживают обнаруживаемую, но умеренную консервацию [16], [19].Мы применили этот же метод к известным функциональным lincRNA человека и обнаружили, что большинство lincRNAs, идентифицированных в этом исследовании, демонстрируют уровень консервации, соответствующий известным функциональным lincRNAs (рис. 3C).
LincRNA обогащены SNP, ассоциированными с признаками
Почти половина всех ассоциированных с признаками SNPs (TASs), идентифицированных в исследованиях общегеномных ассоциаций, расположена в межгенной последовательности, в то время как только небольшая часть находится в экзонах генов, кодирующих белок [1]. Это любопытное наблюдение указывает на обилие функциональных элементов в межгенной последовательности.Хотя некоторые из этих областей могут функционировать только на уровне ДНК, возможно, что многие из них функционируют, кодируя РНК. Фактически, ТАС уже были идентифицированы внутри или рядом с некодирующими РНК, включая некоторые lincRNAs [16], [33] — [36]. Мы рассудили, что если lincRNAs функциональны, они должны быть обогащены TAS по сравнению с неэкспрессируемыми межгенными регионами. Действительно, мы обнаружили, что lincRNAs более чем в 5 раз обогащены TAS по сравнению с неэкспрессируемыми межгенными регионами (Рисунок 4), несмотря на примерно равное распределение SNP между этими регионами (Рисунок S7).Следовательно, многие межгенные области, ассоциированные с признаками, могут функционировать, кодируя lincRNAs.
Рисунок 4. LincRNAs обогащены SNP, ассоциированными с признаками.
Сравнивается количество ассоциированных с признаком SNP в экзонах гена RefSeq NM, экзонах lincRNA или фоновых локусах (неэкспрессируемая межгенная последовательность) на каждый протестированный SNP в исследованиях полногеномной ассоциации (см. Методы). * P = 0,0173, ** P <2,2E-16; планки ошибок представляют собой 95% доверительный интервал биномиальной пропорции.
https://doi.org/10.1371/journal.pgen.1003569.g004
Discussion
В последнее время велись дебаты о том, существует ли всепроникающая транскрипция генома человека и каково количество и распространенность межгенных транскриптов [9] — [12]. До недавнего времени ключевым отсутствующим компонентом в этой дискуссии был анализ сверхглубоких данных РНК-секвенирования, взятых из широкого спектра типов тканей. Без этого недостаточная глубина считывания может привести к неспособности идентифицировать межгенные транскрипты с низким содержанием, а ограниченный отбор образцов ткани приводит к пропущенной тканеспецифической экспрессии.В ходе этого исследования проект ENCODE выпустил крупномасштабный анализ данных RNA-seq, который предоставил четкие доказательства того, что геном человека транскрибируется повсеместно [14]. Мы проанализировали отдельный дополнительный набор данных последовательности РНК, который также удовлетворяет этим требованиям глубины считывания и ширины ткани, охватывая как полиаденилированные, так и неполиаденилированные фракции РНК. В полном согласии с результатами ENCODE, мы наблюдали, что приблизительно 85% генома транскрибируется, что подтверждает предыдущие наблюдения всеобъемлющей транскрипции, основанные на мозаичных массивах, которые недавно подвергались сомнению [2] — [5].
Существует очевидное несоответствие между этой наблюдаемой всеобъемлющей транскрипцией и относительной малочисленностью аннотированных lincRNAs, наиболее многочисленных межгенных РНК. Следует ожидать, что межгенные области кодируют гораздо больше lincRNAs, чем аннотировано в настоящее время. Действительно, здесь мы обнаружили, что существует намного больше lincRNA, чем было известно ранее, даже после агрессивной фильтрации, которая удалила подавляющее большинство ранее аннотированных длинных некодирующих РНК и недавно обнаруженных межгенных транскриптов (Dataset S2).Эти наблюдения ясно демонстрируют, что человеческий геном транскрибируется повсеместно и что lincRNA составляют чрезвычайно распространенный класс межгенных транскриптов.
В соответствии с предыдущими наблюдениями за меньшими наборами аннотаций lincRNA, наш анализ расширенного каталога lincRNA, представленного здесь, показал, что большинство lincRNAs экспрессируются на более низких уровнях, чем гены, кодирующие белки [16], [19]. Хотя большинство lincRNAs экспрессируются только в нескольких копиях на клетку, мы обнаружили, что многие lincRNAs высоко экспрессируются: около 4000 экспрессируются при> FPKM 10 и почти 1000 экспрессируются при> FPKM 30, конкурируя с экспрессией многих матричных РНК.Мы решили применить ограничение экспрессии, чтобы удалить транскрипты с очень низкой экспрессией из каталога lincRNA. Однако может случиться так, что существует много функциональных lincRNA с очень низкими уровнями экспрессии, ниже порогового значения нашего фильтра экспрессии. Например, функциональная человеческая lincRNA HOTTIP экспрессируется примерно в одной из трех клеток [37]. Более того, недавние открытия показали, что межгенный транскриптом может быть значительно более сложным, чем принято сейчас во внимание, когда рассматриваются транскрипты с очень низкой экспрессией [7].Возможно, что некоторые из них являются функциональными транскриптами, несмотря на их очевидную низкую экспрессию, возможно, имеющие короткие всплески экспрессии во время стадий клеточного цикла или функционирующие в отдельных клетках в гетерогенной популяции, как наблюдалось ранее [14]. Следовательно, хотя мы предоставили наиболее полный каталог lincRNA на сегодняшний день, могут быть дополнительные низкоэкспрессируемые, но потенциально функциональные lincRNA, которые были здесь исключены.
Чтобы свести к минимуму любое возможное загрязнение каталога lincRNA транскриптами, кодирующими белок, использованный подход фильтрации был очень агрессивным.Фактически, большинство ранее аннотированных некодирующих РНК не прошли наши фильтры и поэтому были исключены из каталога lincRNA (таблица S3 и набор данных S9). Подавляющее большинство этих транскриптов (включая большинство «lincRNAs» и «процессированных транскриптов» GENCODEv6) перекрывают известные или предсказанные гены, кодирующие белок, псевдогены или некодирующие РНК, отличные от lincRNA (например, микроРНК) (Таблица S3). Некоторые из этих удаленных транскриптов могут быть функциональными длинными некодирующими РНК, такими как GAS5 (удален, поскольку он содержит 10 генов мяРНК в своих интронах).Однако, чтобы наиболее уверенно идентифицировать только lincRNA, а не потенциальные неаннотированные расширения известных генов, они были удалены.
Из тех ранее аннотированных некодирующих РНК, которые являются межгенными, более половины содержат предсказанные ORF длиной более 100 аминокислот. Например, было обнаружено, что две ранее охарактеризованные функциональные lincRNA человека содержат ORF длиной более 100 аминокислот, Xist и HOTAIR. Эти результаты демонстрируют, что наш подход к фильтрации, который удаляет все транскрипты с открытыми рамками считывания более 100 аминокислот, возможно, позволил удалить некоторые линкРНК с большими нефункциональными рамками считывания.Однако использование отсечки ORF из 100 аминокислот, обычно используемого порога для определения потенциальных генов, кодирующих белок, оправдано, потому что ORF такого размера редко возникают случайно и вместо этого указывают на потенциальную способность кодирования белка [38], [39].
Вместо того, чтобы отбрасывать все транскрипты с большими ORF, как мы сделали здесь, один из вариантов различения транскриптов, которые кодируют, от некодирующих, состоит в анализе частоты замен синонимичных кодонов (PhyloCSF) [40]. Однако этот подход ограничен открытыми рамками считывания, которые могут быть согласованы для разных видов, потенциально пропуская недавно развившиеся или иным образом неконсервативные гены, кодирующие новые белки.Важно отметить, что наш подход к удалению всех транскриптов с большими открытыми рамками считывания эффективно удалял транскрипты со значительным предсказанным кодирующим потенциалом (рис. 2C), указывая на то, что использование ограничения размера ORF по крайней мере так же консервативно, как фильтрация на основе анализа PhyloCSF. Отсутствие вовлечения рибосомы, наблюдаемое с данными рибосомного профилирования, подтверждает строгость отсекающего фильтра ORF (Рисунок 2B). Дальнейший анализ этих удаленных больших межгенных транскриптов, содержащих ORF, выходит за рамки этого исследования, но мы включили эти аннотации для исследователей, заинтересованных в дальнейшем анализе их кодирующего потенциала в поисках новых генов, кодирующих белок (набор данных S10).
Несмотря на то, что большинство ранее аннотированных некодирующих РНК не смогли пройти наши фильтры, наш каталог lincRNA содержит значительно больше lincRNA, чем было известно ранее (> 94% lincRNAs совершенно новые на каждом уровне экспрессии). Это результат двух уникальных особенностей нашего исследования. Во-первых, глубина считывания RNA-seq и разнообразие исследуемых тканей позволили обнаружить редкие и тканеспецифичные транскрипты, которые ранее были неизвестны. Многие из этих новых транскриптов прошли все фильтры и аннотированы как новые lincRNA в нашем каталоге.Во-вторых, в отличие от предыдущих усилий по аннотации lincRNA, которые ограничивались идентификацией только сплайсированных или полиаденилированных lincRNA [16], [19], [41], мы стремились создать аннотации более полного набора lincRNA человека независимо от сплайсинга или полиаденилирования. положение дел. Причины использования такого подхода разнообразны. Две из наиболее известных и распространенных функциональных lincRNA человека, NEAT1 и MALAT1, представляют собой гены с одним экзоном (как и примерно 5% генов, кодирующих белок) [42], указывая тем самым, что несращенные транскрипты могут составлять важный класс lincRNA.Кроме того, описаны многочисленные функциональные неполиаденилированные некодирующие РНК [30], [43]. Даже длинные некодирующие РНК, которые могут быть сплайсированы, часто встречаются в их необработанных формах [44], отличное свойство длинных некодирующих РНК, которое привело бы к пропущенным lincRNAs, если бы сплайсинг был обязательным атрибутом. Поэтому мы решили не исключать какие-либо линкРНК из этого каталога из-за отсутствия сплайсинга или полиаденилирования. Важно отметить, что, поскольку несплицированные, неполиаденилированные транскрипты теоретически могут быть ошибочно собраны de novo из считываний, полученных из загрязненной геномной ДНК в данных РНК-seq, мы приняли несколько мер, чтобы уменьшить любой вклад загрязняющих считываний геномной ДНК (см. Методы).
Из-за неотъемлемых ограничений сборки транскриптома de novo с использованием коротких чтений конечной глубины не всегда возможно однозначно определить полную структуру транскрипта. Это особенно верно для низкоэкспрессируемых транскриптов, где количество доступных чтений ограничено, и для геномных областей, в которых чтения не могут быть однозначно картированы. В случае малой глубины считывания экзоны мультиэкзонных транскриптов могут не иметь считываний, соединяющих экзоны, и сборка de novo может приводить к отдельной аннотации каждого экзона как отдельного транскрипта.В подтверждение этого мы обнаружили, что более низко экспрессированные lincRNAs, обнаруженные из сборки транскриптов de novo , с меньшей вероятностью имеют мультиэкзонные структуры (Таблица S5). Кроме того, аннотированные 5′- и 3′-концы lincRNA могут представлять собой усечения полноразмерных транскриптов. Действительно, наш анализ данных ПЭТ-тегов показал, что, хотя большая часть нашего каталога lincRNA перекрывается по крайней мере одной ПЭТ-тегом, в большинстве случаев существует минимальная поддержка ПЭТ-тегов для аннотированных 5′- и 3′-концов lincRNA (Таблица S6 ).Поэтому некоторые аннотации lincRNA в каталоге, который мы предоставляем (Dataset S2), особенно аннотации lincRNA с одним экзоном, могут представлять собой фрагменты более крупных транскриптов.
Более того, учитывая сообщенную распространенность низкоуровневых перекрывающихся транскриптов по всей межгенной последовательности [7], неясно, могут ли полные структуры lincRNA быть однозначно деконволютированными с использованием технологии короткого считывания RNA-seq. Определение полных структур линкРНК станет важным будущим усилием в этой области и может основываться на новых наборах данных с большей длиной чтения и большей глубины чтения, использовании нескольких ортогональных типов данных в одной и той же ткани, новых технологиях, таких как сверхдлинное считывание следующего поколения. секвенирование и дальнейшие улучшения программного обеспечения для сборки транскриптов de novo .
Кроме того, в большинстве проанализированных нами данных RNA-seq отсутствует информация о цепочке, и в результате большинство lincRNA в нашем каталоге имеют неоднозначную цепочку. Предыдущие аннотации полагались на ориентацию сайта сплайсинга, чтобы сделать вывод о многонитевой структуре транскрипта [16]. Хотя это разумный подход, который мы также приняли, когда он применим в настоящем каталоге lincRNA, данные о цепочечной РНК-seq необходимы для наиболее уверенного определения цепочечности для собранных de novo транскриптов.
Хотя определение изоформ и полных структур всех lincRNAs явно желательно, эти неполные аннотации структуры lincRNA, тем не менее, имеют огромное практическое значение. Знания структуры части транскрипта часто достаточно для проверки дифференциальной экспрессии или проведения экспериментов по нокдауну РНКи, а также для облегчения клонирования и секвенирования полноразмерного транскрипта. Из-за этого, вместо того, чтобы накладывать дополнительные ограничения на аннотации lincRNA, наша стратегия фильтрации была направлена на идентификацию как можно большего количества транскриптов, которые соответствуют определению lincRNA.Однако для исследователей, заинтересованных в более точных аннотациях lincRNA, мы предоставили несколько более ограничительных каталогов lincRNA (Datasets S4, S5, S6).
Ключевой вопрос в этой области — являются ли транскрипты, полученные в результате всепроникающей транскрипции межгенных областей, функциональными или являются результатом шумной транскрипции. Описанные нами lincRNA специфически регулируются и содержат консервативные последовательности, атрибуты которых несовместимы с шумом транскрипции (рис. 3). Кроме того, было обнаружено, что lincRNAs сильно обогащены межгенными TAS по сравнению с неэкспрессируемыми межгенными регионами (Рисунок 4).Это поразительное открытие подтверждает возможность того, что многие межгенные SNP маркируют области, которые функционируют как lincRNAs, а не как элементы ДНК. Поскольку почти половина всех TAS являются межгенными, возможно, что lincRNA играют значительную роль в большинстве человеческих черт и заболеваний, проанализированных на данный момент в GWAS. Одна функциональная lincRNA (MIAT) была впервые идентифицирована во время экспериментального исследования межгенного TAS [35], а другая lincRNA PTCSC3 была идентифицирована рядом с TAS, обнаруженным из папиллярной карциномы щитовидной железы GWAS, что, возможно, представляет собой первое из многих таких открытий, которые были сделаны межгенные ТАС.Обнаружение того, что lincRNAs сильно обогащены TASs, предоставляет новую возможность пересмотреть области, ассоциированные с межгенными признаками, с неизвестными функциональными механизмами, проверяя, участвует ли перекрывающаяся lincRNA в наблюдаемом фенотипе.
Этот каталог некодирующих РНК представляет собой важный шаг на пути к более полному пониманию этого захватывающего рубежа. Мы идентифицировали большое количество предполагаемых lincRNAs с характеристиками, предполагающими функциональность. Однако многие из этих lincRNA слабо экспрессируются, и окончательное доказательство функциональности lincRNA требует функциональных экспериментов.Высокопроизводительные функциональные геномные подходы, такие как скрининг сверхэкспрессии РНКи и кДНК, будут служить важнейшими инструментами для будущих усилий по раскрытию роли линкРНК в различных биологических системах. С необходимой технологией, доступной сейчас для этих экспериментальных подходов следующего поколения, настало время, чтобы эта темная материя человеческого генома еще больше вышла в центр внимания.
Материалы и методы
РНК-seq и профилирование рибосом читать выравнивание и обработка
127 файлов последовательностей RNA-seq (5 новых и 122 общедоступных набора данных, таблица S1) были выровнены по hg18 с помощью TopHat v1.1.4 разрешает только однозначно отображенные чтения с использованием опции -g 1 (все остальные параметры были по умолчанию, см. Руководство TopHat http://tophat.cbcb.umd.edu/manual.html). Подробная информация, относящаяся к каждому набору данных, включая новые наборы данных, доступна в источниках, представленных в таблице S1. Эти наборы данных RNA-seq были выбраны потому, что они охватывают широкий спектр человеческих тканей и типов клеток, имеют хорошо задокументированные экспериментальные методы, используемые для их создания, и являются общедоступными. В то время как наборы данных с более длинными чтениями и большей глубиной чтения были предпочтительнее, поскольку они позволяют более полную сборку транскриптов de novo , некоторые наборы данных с короткими чтениями и малой глубиной чтения были включены, чтобы отобрать образцы как можно большего количества типов тканей.Наборы данных, полученные из тканей с мутировавшими геномами, такими как рак, были включены для захвата тканеспецифической экспрессии, даже если некоторые считывания из мутированных геномных позиций не могли отображаться в эталонном геноме hg18. SAMtools v0.1.7 и BEDTools v2.12.0 использовались для обработки выровненных файлов чтения.
Количественное определение транскрибированной фракции генома
Уникально отображаемый геном человека, определяемый здесь как части генома, в которые могут быть однозначно сопоставлены чтения RNA-seq, был получен для hg18 из http: // www.imagenix.com/uniqueome/downloads/hg18_uniqueome.unique_starts.base-space.50.2.positive.BED.gz [45]. Он содержит 2 570 174 327 п.н. или 83,4% всей геномной последовательности человека. Чтобы определить геномный охват данных RNA-seq, все выровненные считывания RNA-seq были объединены, и охват считыванием в каждой позиции геномного основания был определен с помощью функции genomeCoverageBed BEDTools. Расщепленные чтения (т.е. считывания, охватывающие соединение экзон-экзон) подсчитывались таким образом, чтобы интронная последовательность была включена как часть считывания.На рисунке 1A «Все гены, EST, кДНК» включают гены GENCODE v10 (за исключением псевдогенов), гены RefSeq NM и NR, известные гены UCSC, сплайсированные кДНК H-Invitational, сплайсированные EST (трек UCSC Genome Browser «Spliced EST»), и ранее аннотированные сплайсированные lincRNAs [16]. Во всех случаях были включены интронные последовательности генов, кДНК и EST.
Открытие LincRNA
Было собранотранскриптов, аннотированных в общедоступных базах данных и литературных источниках, которые могли быть линкРНК.
Ensembl v61 категории «обработанный_транскрипт» и «lincRNA», категории GENCODE v6 «обработанный_транскрипт» и «lincRNA», гены RefSeq NR и XR, «некодирующие» транскрипты H-Invitational, ультраконсервативные элементы (UCE) и опубликованные lincRNA из Khalil et al. [18] и Cabili et al. [16]. LiftOver использовался для сопоставления координат hg19 с hg18 для Ensembl, GENCODE, H-Invitational и Cabili et al. [16] расшифровок. Последовательности RefSeq XR в hg19 были выровнены с hg18 с помощью BLAT v34, и было использовано выравнивание с наивысшей оценкой. Последовательности ультраконсервативных элементов были извлечены из http://biodev.cbm.fvg.it, выровнены по hg18 с помощью BLAT v34, и было использовано выравнивание с наивысшей оценкой. Халил и др. [18] экзонов были сгруппированы по их перекрывающимся определенным транскрибируемым областям для построения структур транскриптов.
Были собраны новые транскрипты из
de novo сборки транскриптома данных РНК-seq.Сборка транскриптома De novo была выполнена на данных РНК-seq с Cufflinks v1.0.1 с использованием параметров нормализации верхнего квартиля (-N) и коррекции смещения фрагментов (-b). Сборка этого транскрипта была выполнена с использованием считываний, которые были предварительно выровнены по hg18 с использованием TopHat, как описано выше. В случаях, когда было доступно несколько наборов данных одного и того же типа библиотеки из одной и той же ткани, эти наборы данных были объединены для увеличения глубины чтения для сборки de novo (см. Таблицу S2).Для парных наборов данных конечного чтения использовались только правильно парные и одноэлементные чтения, как определено SAMTools.
Транскрипты были отфильтрованы для удаления перекрытия с генами, не относящимися к lincRNA, или псевдогенами и короткими транскриптами.
Были удалены транскрипты длиной менее 200 нуклеотидов. Остальные транскрипты удаляли, если они находились в пределах 1 т.п.н. от генов RefSeq NM на той же цепи или, в случае транскриптов с неоднозначной цепочкой, на любой цепи относительно гена NM. Транскрипты на противоположной цепи гена NM удаляли, если они перекрывали ген NM по крайней мере на одно основание.Кроме того, были удалены транскрипты, перекрывающиеся по крайней мере с одним основанием любого из следующих, независимо от многонитевой: гены Ensembl v61, кроме «lincRNA» и «loaded_transcript», нечеловеческие гены RefSeq, выровненные по hg18 с помощью BLAT (UCSC Genome Browser «Other RefSeq «Трек»), альтернативные и расширенные 5 ‘и 3’ UTR известных человеческих генов из транскриптов UTRdb, RefSeq NR и XR, аннотированных как «псевдогены», и кодирующих последовательностей Ensembl v54.
Было удаленотранскриптов, содержащих большие ORF.
Были выполнены два этапа фильтрации для удаления предполагаемых транскриптов, кодирующих белок, и их UTR. Во-первых, с помощью EMBOSS getorf v6.1.0 были идентифицированы большие ORF (> 100 аминокислот) во всех транскриптах во всех рамках считывания. Чтобы учесть потенциально усеченные транскрипты, содержащие ORF, в которых стартовый или стоп-кодон может находиться за пределами аннотированной области, наличие более 300 нуклеотидов ниже стартового кодона без прерывающего стоп-кодона или 300 нуклеотидов перед стоп-кодоном кодона без прерывающего стартового кодона, достаточного для вызова предполагаемой ORF.Транскрипты с предполагаемыми большими ORF были удалены. Эти предполагаемые большие ORF, содержащие межгенные транскрипты, некоторые из которых могут быть новыми генами, кодирующими белок, предоставляются в качестве ресурса в Dataset S10. Чтобы удалить потенциальные UTR этих больших ORF-содержащих транскриптов из каталога lincRNA, оставшиеся транскрипты были отфильтрованы, чтобы удалить любые, которые перекрывают большой транскрипт, содержащий ORF.
Было удаленотранскриптов, перекрывающих расширенные структуры генов, кодирующих белок.
РНК-seq чтения могут выходить за аннотированные 5′- и 3′-концы аннотированных структур генов, кодирующих белок, представляющих возможные протяженные UTR, а также, в случае картирования сплайсированных считываний на ген из дистальных сайтов, неаннотированных экзонов. Чтобы избежать каталогизации транскриптов в этих областях как lincRNA, мы создали фильтр, основанный на расширенных границах генов, кодирующих белок, с использованием данных RNA-seq. Для этого сборка транскриптома de novo с помощью Cufflinks v1.1.0 с использованием генов RefSeq NM в качестве справочной аннотации (-g), нормализация верхнего квартиля (-N) и коррекция смещения фрагментов (-b) выполнялись для всех полиA + РНК. -seq библиотеки в таблице S2.Аннотации гена RefSeq NM использовались в качестве справочной аннотации для этой сборки транскриптов, поскольку они представляют собой ограниченный набор аннотаций генов, кодирующих белок с высокой степенью достоверности. Этот набор границ расширенного гена, кодирующего белок (набор данных S1), использовали в качестве фильтра для удаления транскриптов, которые перекрывают любой ген, кодирующий расширенный белок, по крайней мере на одно основание, независимо от цепочки.
Транскрипты, не экспрессирующиеся при FPKM> 1 по крайней мере в одном наборе данных, были удалены.
Чтобы определить уровни экспрессии транскриптов, картированные чтения РНК-seq распределяли по транскриптам с использованием модифицированной версии HTSeq v0.5.3p, который позволяет считывать чтения, сопоставленные с разделяемыми частями перекрывающихся транскриптов, как полное чтение для каждой перекрывающейся транскрипции. Это было необходимо для правильного присвоения чтения каждой из нескольких избыточных аннотаций транскриптов, присутствующих в объединенном наборе из всех общедоступных баз данных и сборок de novo до слияния перекрывающихся аннотаций lincRNA (описано ниже). Эти избыточные аннотации являются результатом повторной сборки de novo одной и той же транскрипции в нескольких различных наборах данных или избыточных существующих аннотаций в общедоступных базах данных, каждая из которых имеет немного разные геномные координаты, но может представлять одну и ту же транскрипцию.Таким образом, все чтения были полностью распределены по каждой избыточной аннотации, а не пропорционально между ними. Счетчики чтения были преобразованы в FPKM с использованием общего числа отображенных операций чтения для каждого набора данных, рассчитанного функцией SAMTools flagstat и пользовательскими скриптами. Транскрипты, не выраженные при FPKM> 1 по крайней мере в одном наборе данных, были удалены. В результате этого минимального фильтра FPKM> 1, 99.975% из de novo собранных lincRNAs (до слияния) имеют по крайней мере 5 чтений, поддерживающих их экспрессию по крайней мере в одном из объединенных наборов данных в таблице S2, и> 99.1% имеют не менее 10 чтений в одном наборе данных. Транскрипты были дополнительно классифицированы как FPKM> 1, FPKM> 10 и FPKM> 30 по крайней мере в одном наборе данных, где каждая категория включает все транскрипты в более высоких категориях.
Перекрывающиеся транскрипты, прошедшие все фильтры на каждом пороге экспрессии, были объединены и сгруппированы по близости.
Чтобы идентифицировать минимальный набор различных lincRNA, перекрывающиеся транскрипты объединяли, если 50% экзона транскрипта перекрывали экзон другого транскрипта.Кроме того, слитые транскрипты в пределах 1 т.п.н. друг от друга были помещены в одну и ту же группу, но получили разные номера транскриптов и названы на основе уровня экспрессии FPKM, из которого они были получены, например FPKM1_group_32871_transcript_1. Слияние, группирование и именование выполняли отдельно для всех транскриптов FPKM> 1, транскриптов FPKM> 10 и транскриптов FPKM> 30. Статистика фильтрации представлена в таблице S3. Каталог слитых lincRNA на каждом отрезке экспрессии находится в формате BED для построения генома hg18 в Dataset S2.Каталог линкРНК FPKM> 1 использовался для всех анализов в этом исследовании, если не указано иное. Аннотации lincRNA представлены в виде файлов BED в аннотации генома hg18, а не hg19, потому что браузер генома UCSC в настоящее время имеет больше «треков» данных, доступных для hg18. Однако аннотации lincRNA могут быть легко преобразованы в hg19 или другие аннотации генома пользователями с помощью инструмента LiftOver: http://genome.ucsc.edu/cgi-bin/hgLiftOver.
После объединения этих фильтрованных экспрессий, перекрывающихся lincRNA, FPKM были пересчитаны (Dataset S8) для объединенных lincRNA с использованием модифицированной программы HTSeq, описанной выше.Из-за неполной природы структур lincRNA, возникающих в результате сборки de novo , перекрывающиеся и соседние lincRNAs считались представляющими разные потенциальные модели одного и того же гена lincRNA (а не изоформ). Следовательно, в редких случаях, когда две или более модели lincRNA частично перекрываются, но не удовлетворяют нашим критериям слияния (выше), картирование чтения на эти перекрывающиеся части было полностью назначено каждой lincRNA.
Идентификация lincRNAs, экспрессируемых значительно выше других межгенных областей.
Для каждого набора данных RNA-seq (таблица S1) было сгенерировано эмпирическое фоновое распределение значений экспрессии с использованием одного миллиона аннотаций с подобранным размером, случайным образом перемешанных по межгенной последовательности. Используемая межгенная последовательность включает все части уникально картируемого генома, за исключением генов RefSeq NM, NR и XR, генов Ensembl v61, включая «lincRNA» и «процессированные транскрипты», гены GENCODEv6, включая «lincRNA» и «процессированные транскрипты», H-Invitational « некодирующие »транскрипты, альтернативные и расширенные 5 ‘и 3’ UTR известных человеческих генов из UTRdb, расширенные структуры генов, кодирующих белок, полученные из данных РНК-seq (расширенный фильтр генов, описанный выше), и опубликованные lincRNA из Khalil et al. [18] и Cabili et al. [16]. Чтобы определить, какие предполагаемые lincRNA (в наборе данных S2, FPKM> 1) были выражены значительно выше фона, по крайней мере, в одном наборе данных, вероятность наблюдения транскрипта на любом заданном уровне экспрессии была оценена с использованием фонового распределения для конкретного набора данных и скорректирована для нескольких тестов в соответствии с с поправкой Бонферрони, предполагающей один тест на набор данных RNA-seq. Эти аннотации lincRNA с исправленным значением P <= 0.1 по крайней мере в одном наборе данных каталогизированы в наборах данных S6, S7.
дополнительных LincRNA, экспрессируемых только у Cabili et al. [16]
Дополнительный набор аннотированных транскриптов lincRNA от Cabili et al. [16] прошел все наши фильтры, за исключением того, что они не были выражены при FPKM> 1 ни в одном из анализируемых здесь наборов данных, и поэтому были удалены из каталога lincRNA в наборе данных S2. Однако сообщалось, что некоторые из этих транскриптов выражены при FPKM> 1 по крайней мере в одном из наборов данных, проанализированных в Cabili et al. [16], все они отличаются от анализируемых здесь наборов данных. Эти дополнительные lincRNA были объединены с lincRNA в каталоге в Dataset S2, что привело к дополнительным 920 lincRNA в 741 группе при FPKM> 1, 88 lincRNAs в 82 группах при FPKM> 10 и 17 lincRNAs в 17 группах при FPKM> 30. Этот расширенный каталог lincRNA находится в формате BED для построения генома hg18 в наборе данных S3 и не использовался в дальнейшем для каких-либо анализов в этом исследовании.
Примечание о загрязнении геномной ДНК в наборах данных RNA-seq
Загрязнение геномной ДНК является потенциальным источником ложноположительного сигнала экспрессии в данных РНК-seq, который может способствовать сборке de novo ошибочных транскриптов.В принципе, только считывания, охватывающие соединение экзон-экзон, могут быть однозначно определены как происходящие от РНК. Следовательно, правильная сборка de novo как несплайсированных, так и сплайсированных (помимо считываний, охватывающих соединения экзон-экзон) транскриптов может пострадать, если присутствует значительная контаминация геномной ДНК. Поскольку в нашем анализе использовался широкий спектр новых и ранее существовавших наборов данных RNA-seq с неизвестным содержанием контаминации геномной ДНК, мы предприняли несколько шагов, чтобы уменьшить эту возможность.Во-первых, для всех наборов данных RNA-seq мы проанализировали распределение считываний между экзонами, кодирующими белок, по сравнению с другими регионами, ожидая, что распределения чтения должны быть аналогичными между наборами данных RNA-seq, созданными из библиотек одного типа (например, выбранный polyA +). Набор данных с необычно высоким процентом интронных и межгенных считываний может содержать значительную контаминацию геномной ДНК. Наш анализ наборов данных, используемых в этом исследовании, показал, что, как и ожидалось, наборы данных, специфичных для полиА + РНК-seq, имеют более высокую долю считываний, отображаемых на экзоны генов, кодирующих белок, чем наборы данных с истощенной рРНК или полиА-.Более того, не было обнаружено явных наборов данных с выбросами ни для одного из типов библиотек. Результаты этого анализа подтвердили, что в этом исследовании не использовались наборы данных с высоким содержанием геномной ДНК (рис. S2). Затем, как описано на рис. 2А и в разделе «Методы», мы применили пороговые значения как по размеру, так и по экспрессии для всех линкРНК. Ограничение по размеру предотвращает ошибочное считывание ошибочных одиночных считываний либо из-за загрязнения геномной ДНК, либо из-за артефактов картирования считывания как lincRNA, в то время как ограничение экспрессии удаляет lincRNA, которые собираются из редких считываний, полученных из геномной ДНК.Комбинация этих подходов позволила минимизировать вклад геномной ДНК в каталог lincRNA.
Анализ распределения LincRNA между полиаденилированной и неполиаденилированной РНК-seq данными
Были проанализированы данныеH9 ESC и HeLa RNA-seq из фракций, содержащих исключительно полиА- или полиА + транскрипты [46]. Транскрипты с считыванием RNA-seq во всех четырех наборах данных и с FPKM> 1 по крайней мере в одной из двух фракций для каждого типа клеток были проанализированы для рисунка 3B (16 819 генов NM и 127 lincRNA).Для рисунка S5 транскрипты с показаниями в обеих фракциях и FPKM> 1 в по крайней мере одной из двух фракций для определенного типа клеток были включены в анализ этого типа клеток (20 470 NM генов и 849 lincRNA в ESC H9; 18 294 NM генов. и 1009 линкРНК в HeLa). Усы прямоугольника и усов простираются в +/- 1,5 раза от межквартильного размаха или самой экстремальной точки данных.
Кластерный анализ парных цифровых тегов (ПЭТ)
Общедоступные кластерные аннотации парных тегов (PET), полученные из 7 клеточных линий или тканей, созданные в рамках проекта ENCODE, были загружены с http: // genome.ucsc.edu/cgi-bin/hgFileUi?db=hg19&g=wgEncodeGisRnaPet. Использовались файлы аннотаций кластера ПЭТ (по типу клеток или тканей):
A549 (wgEncodeGisRnaPetA549CellPapClusters.bedCluster),
h2_hESC (wgEncodeGisRnaPeth2hescCellPapClustersRep1.bed),
HeLa-S3 (wgEncodeGisRnaPetHelas3CellPapClustersRep1.bed),
IMR90 (wgEncodeGisRnaPetImr90CellPapClusters.bedCluster),
MCF-7 (wgEncodeGisRnaPetMcf7CellPapClusters.bedCluster),
Простата (wgEncodeGisRnaPetProstateCellPapClustersRep1.кровать),
СК-Н-Ш (wgEncodeGisRnaPetSknshCellPapClusters.bedCluster).
Дальнейшее описание этих ПЭТ-кластеров, включая способ создания аннотаций, доступно на сайте UCSC Genome Browser здесь http://genome.ucsc.edu/cgi-bin/hgTrackUi?hgsid=321010719&c=chr21&g=wgEncodeGisRnaPet. BEDTools использовался для вычисления перекрытия между lincRNA и 5 ‘и 3’ концами гена RefSeq NM и 5 ‘и 3’ концевыми ‘блоками’ кластера ПЭТ. В случае неоднозначных цепочечных lincRNAs обе потенциальные ориентации были разрешены для определения перекрытия с 5′- и 3′-концами PET-кластеров.
Анализ профиля рибосом
Данные профилирования рибосом и сопоставленные данные мРНК-seq из клеток HeLa, соответствующие экспериментам (фиктивная трансфицированная точка времени 12 часов), представленные в Guo et al. [22] были загружены из NCBI GEO (GSE22004). Уровень экспрессии отфильтрованного набора lincRNA и транскриптов RefSeq NM оценивали, как указано выше. 803 lincRNA, экспрессируемые при FPKM> 1, и образец из 1292 транскриптов RefSeq NM, экспрессируемых при FPKM> 1 (разделенных на составляющие их области CDS и 3′-UTR), были разбиты на окна 30 п.н. со смещением 1 п.н.Модифицированная версия HTSeq (описанная выше) использовалась для подсчета считываний, выравнивающихся по каждому окну, как для данных RNA-seq, так и для данных рибосомного профилирования. Соотношение считываний, связанных с рибосомами, и считываний последовательностей мРНК оценивали для каждого окна, и максимальное соотношение для данного транскрипта принимали как меру взаимодействия с рибосомами. «Усы» прямоугольника и «усов» на рис. 2В простираются в +/- 1,5 раза от межквартильного размаха с выбросами, изображенными точками. Для вычисления значений P использовали критерий суммы рангов Вилкоксона.
Вычислительный анализ возможностей кодирования
Программа PhyloCSF (выпуск от 16.09.2010) [40] была использована для вычислительной оценки кодирующего потенциала отфильтрованных транскриптов lincRNA. BED-файл, описывающий эти транскрипты lincRNA, а также случайную выборку 8310 транскриптов RefSeq NM был загружен на веб-сервер Galaxy (https://main.g2.bx.psu.edu/) и использовался инструмент Stitch Gene Blocks. чтобы получить несколько файлов выравнивания с записями последовательности для следующего генома сборок на основе 44 пути выравнивания Multiz к hg18: hg18 panTro2 rheMac2 tarSyr1 micMur1 otoGar1 tupBel1 ММ9 RN4 dipOrd1 cavPor3 speTri1 oryCun1 ochPri2 vicPac1 turTru1 bosTau4 equCab2 felCat3 canFam2 myoLuc1 pteVam1 eriEur1 sorAra1 loxAfr2 proCap1 echTel1 dasNov2 choHof1.Имена сборки генома были преобразованы в общие имена, и PhyloCSF запускался с использованием параметров –orf = StopStop3 и –frames = 6.
Анализ модификации хроматина
ДанныеChIP-seq из ячеек IMR90 [28] были извлечены из NCBI SRA (Таблица 1) и выровнены с hg18 с помощью Bowtie v0.12.7, разрешающей только однозначно отображаемые чтения (-k 1). Модифицированная версия HTSeq v0.5.3p (описанная выше) использовалась для подсчета карт считывания с lincRNAs и RefSeq NM генами. Отношение считываний IP к совпадающим считываниям входного контроля использовалось в качестве меры сигнала ChIP.Данные РНК-seq из клеток IMR90 [29] также были проанализированы для получения значений FPKM для lincRNAs и RefSeq NM генов с использованием той же процедуры, что и для обнаружения lincRNA. «Усы» на графике «прямоугольник» и «усы» простираются в +/- 1,5 раза от межквартильного размаха или самой экстремальной точки данных.
Кластеризация тканей с помощью экспрессии LincRNA
Наборы данныхRNA-seq из B-клеток, h2 ESC и мозга (см. Таблицу S1) были сгруппированы по уровням экспрессии lincRNA. LincRNA с FPKM> 10 в одном или нескольких из 7 наборов данных RNA-seq, проанализированных на рисунке 3B, были использованы для создания тепловой карты и дендрограммы.Эти 7 наборов данных были выбраны для этого анализа, потому что они имеют реплики из каждой ткани и имеют счетчики глубокого считывания для всех реплик (Таблица S1), важные особенности для точного измерения дифференциальной экспрессии. Используя Gene Cluster 3.0, значения FPKM были преобразованы log 2 , а гены (строки) и образцы (столбцы) были нормализованы путем умножения каждого log 2 преобразованного значения FPKM на масштабный коэффициент, так что сумма квадратов значений в каждой строке и столбце по 1.0. Евклидово расстояние с использованием центроидной связи было рассчитано для всех образцов, а тепловая карта и дендрограмма были созданы с помощью Java TreeView. Красный цвет соответствует полностью индуцированной экспрессии, а синий соответствует полностью подавленной экспрессии.
Анализ сохранения
Базовые оценки сохранения (оценка PhyloP, рассчитанная с помощью PHAST), основанная на множественном выравнивании геномов плацентарных млекопитающих, были загружены из браузера генома UCSC. Было идентифицировано окно 50 п.н. в каждом транскрипте lincRNA с наивысшим средним баллом PhyloP.Процесс повторяли для генов RefSeq NM и набора повторяющихся элементов, совпадающих по размеру (lincRNA), из RepeatMasker (UCSC Genome Browser). Показатели PhyloP для максимально консервативных окон размером 50 п.н. каждой линкРНК приведены в таблице S4.
Анализ SNP
Обогащение SNP, связанных с признаками.
Таблица, содержащая все связанные с признаками SNP с P <10 −8 , была загружена из обозревателя результатов ассоциации dbGaP NCBI (всего 3781 связанный с признаками SNP).Геномные координаты ассоциированных с признаками SNP были получены из dbSNP 130. Чтобы сравнить обогащение ассоциированных с признаками SNP в lincRNA по сравнению с фоновыми локусами (неэкспрессированные межгенные области), области уникально картируемого генома длиннее 200 п.н., которые исключают все доказательства транскрипции (РНК -seq читает, RefSeq NM, гены и псевдогены NR и XR, гены Ensembl v61, гены GENCODE v10, сплайсированные EST, сплайсированные кДНК H-Invitational, 5 ‘и 3’ UTR из UTRdb, расширенные гены RefSeq NM, полученные с использованием справочной аннотации Сборка транскриптома de novo (см. выше и набор данных S1) и все lincRNA) были скомпилированы и служили фоновыми локусами для этого анализа.Количество тестируемых SNP на массивах SNP Illumina (Illumina 1M) и Affymetrix (Affymetrix SNP Array 6.0) определяли для экзонов гена RefSeq NM, экзонов lincRNA и фоновых локусов. Количество тестируемых SNP на платформу было масштабировано путем дробного вклада платформ Illumina (58,6%) по сравнению с Affymetrix (41,4%) в полный набор GWAS в каталоге NHGRI GWAS [1]. Затем с использованием этого масштабированного числа протестированных SNP определяли количество связанных с признаками SNP на каждый протестированный SNP. Для вычисления значений P использовался точный критерий Фишера, и планки ошибок на рисунке 4 представляют собой 95% доверительные интервалы биномиальной пропорции.
Общие SNP.
Таблица, содержащая все распространенные SNP (частота минорных аллелей> 0,05) из версии HapMap № 27 была загружена с сайта BioMart HapMap (http://hapmap.ncbi.nlm.nih.gov/biomart/martview) и количество Были определены общие SNP внутри экзонов гена RefSeq NM, экзонов lincRNA и фоновых локусов, разделенные на количество геномных оснований в каждой из этих категорий. Для вычисления значений P использовался точный критерий Фишера, и планки ошибок на рисунке S7 представляют собой 95% доверительные интервалы биномиальной пропорции.
Вспомогательная информация
Набор данных S8.
RNA-seq FPKM и счетчики чтения для всех lincRNA (из набора данных S2, FPKM> 1) и генов NM во всех отдельных наборах данных (TXT). Обратите внимание, что это большие файлы: сжатый файл FPKM имеет размер 32 МБ (94 МБ без сжатия), а сжатый файл счетчиков — 7 МБ (29 МБ без сжатия).
https://doi.org/10.1371/journal.pgen.1003569.s008
(ZIP)
Набор данных S9.
GENCODEv6 «линкРНК» и «обработанные транскрипты», которые удалялись на каждом этапе фильтрации.(A) Нефильтрованные GENCODEv6 «линкРНК» и «процессированные транскрипты» (39 472 транскрипта) (формат BED; hg18) (TXT). (B) GENCODEv6 «линкРНК» и «процессированные транскрипты», которые перекрывают гены RefSeq NM (кодирующие белки) по крайней мере на 1 пару оснований на каждой цепи (27 267 транскриптов) (формат BED; hg18) (TXT). (C) GENCODEv6 «линкРНК» и «процессированные транскрипты», которые перекрывают (см. Методы) один или несколько элементов расширенного набора генов, кодирующих белок (UCSC, RefSeq, Ensembl, GENCODE), псевдогенов, UTR (UTRdb) или не- lincRNA некодирующие РНК (33 245 транскриптов) (формат BED; hg18) (TXT).(D) GENCODEv6 «линкРНК» и «процессированные транскрипты», которые прошли белок / псевдоген / не линкРНК нкРНК / фильтр <200 нт, но содержат ORF> 100 аминокислот в длину (964 транскрипта) (формат BED; hg18) ( ТЕКСТ). (E) GENCODEv6 «линкРНК» и «процессированные транскрипты», которые сами по себе не содержат ORF> 100 аминокислот, но перекрывают другую аннотированную или de novo линкРНК, которая содержит ORF> 100 аминокислот (2700 транскриптов) (формат BED; hg18) (TXT). (F) GENCODEv6 «линкРНК» и «процессированные транскрипты», которые прошли предыдущие фильтры, но перекрывают расширенную структуру гена, кодирующего белок (149 транскриптов) (формат BED; hg18) (TXT).(G) GENCODEv6 «lincRNAs» и «обработанные транскрипты», прошедшие все предыдущие фильтры, за исключением не обнаруженных экспрессированных при FPKM> 1 ни в одном наборе данных (1469 транскриптов) (формат BED; hg18) (TXT). (H) GENCODEv6 «линкРНК» и «обработанные транскрипты» проходят все фильтры и экспрессируются при FPKM> 1 по крайней мере в одном наборе данных (945 транскриптов) (формат BED; hg18) (TXT).
https://doi.org/10.1371/journal.pgen.1003569.s009
(ZIP)
Рисунок S1.
Фракция человеческого генома с отображенными считывающими последовательностями РНК при различных минимальных порогах считывания.4,5 миллиарда отображенных считываний из всех 127 наборов данных RNA-seq были объединены и сопоставлены с уникально отображаемой частью генома человека (см. Методы). На графике нанесена доля уникально отображаемого генома с минимальным порогом чтения. Данные не выходят на плато при низких минимальных порогах чтения, что указывает на то, что более глубокое секвенирование приведет к дальнейшему увеличению доли покрытого генома. Для разделенных чтений (считываний, охватывающих интрон) промежуточная (интронная) последовательность либо предполагалась как транскрибируемая (включая предполагаемые основания), либо нет (за исключением предполагаемых оснований).При пороговом значении минимального количества считываний в 1 считывание 67,1% и 78,9% генома имеют покрытие считыванием при исключении или включении предполагаемых оснований, соответственно.
https://doi.org/10.1371/journal.pgen.1003569.s011
(TIF)
Рисунок S2.
Фракция считывания RNA-seq, картирование экзонов гена, кодирующего белок (RefSeq NM), в сравнении с интронными и межгенными областями для 127 наборов данных RNA-seq, сгруппированных по типу библиотеки RNA-seq. Подсчет чтения производился с использованием модифицированной версии HTSeq v0.5.3p (см. Методы). Изоформы генов, кодирующих белок, были сглажены перед подсчетом считываний, так что считывания распределялись только один раз на ген, даже если существует несколько изоформ. Библиотеки, отобранные PolyA + (обогащенные мРНК), содержат более высокую фракцию считываний, отображаемых на экзоны генов, кодирующих белок, в то время как библиотеки RNA-seq с обедненной рибосомной РНК и библиотеки, отобранные полиA-, содержат более высокую долю интронных и межгенных считываний. Во всех случаях из-за в целом высоких уровней экспрессии генов, кодирующих белок, экзоны генов, кодирующих белок, содержат непропорционально большое количество картированных считываний по сравнению с занимаемым ими пространством генома (<3%).
https://doi.org/10.1371/journal.pgen.1003569.s012
(TIF)
Рисунок S4.
LincRNAs обладают тканеспецифическими паттернами экспрессии. Уровни экспрессии LincRNA (FPKM) использовали для кластеризации реплик данных РНК-seq из В-клеток, эмбриональных стволовых клеток h2 и ткани мозга. Агломеративная иерархическая кластеризация как линкРНК (строки), так и образцов (столбцы) по евклидову расстоянию была выполнена с log 2 трансформированных значений FPKM lincRNA для lincRNAs с FPKM> 10 по крайней мере в одном из проанализированных образцов.На тепловой карте отображается красный цвет для полностью индуцированных lincRNA и синий для полностью репрессированных lincRNA, где строки и столбцы были нормализованы (см. Методы).
https://doi.org/10.1371/journal.pgen.1003569.s014
(TIF)
Рисунок S5.
Полиаденилирование линкРНК по сравнению с генами, кодирующими белок. Распределение соотношений FPKM во фракциях polyA + / polyA- для lincRNAs и NM генов в ЭСК HeLa и H9. Транскрипты с считыванием в обеих фракциях и FPKM> 1 по крайней мере в одной из двух фракций для определенного типа клеток были включены в анализ этого типа клеток (20 470 генов NM и 849 lincRNA в ESCs H9; 18 294 гена NM и 1009 lincRNA в HeLa).Усы простираются до +/- 1,5 межквартильного размаха или до наиболее экстремальной точки данных.
https://doi.org/10.1371/journal.pgen.1003569.s015
(TIF)
Рисунок S6.
Сравнение сохранности полного каталога lincRNA (53 864 lincRNA, набор данных S2, FPKM> 1) с lincRNA GENCODEv6. Определяли максимально консервативные окна размером 50 п.н. в каждом lincRNA, гене RefSeq NM и повторяющемся элементе (неконсервативные контрольные последовательности). Оценивались только линкРНК GENCODE, прошедшие все фильтры линкРНК (2414 линкРНК GENCODE, таблица S3).
https://doi.org/10.1371/journal.pgen.1003569.s016
(TIF)
Рисунок S7.
Распределение общих SNP между экзонами lincRNA, экзонами гена NM и неэкспрессируемыми межгенными областями. Подсчитывали SNP HapMap II с частотой минорных аллелей> 0,05, расположенные внутри экзонов гена NM, экзонов lincRNA или фоновых локусов (неэкспрессированные межгенные области), нормализованные по общему количеству пар оснований в каждой области (* P = 0,0173, ** P <2,2E-16; полосы ошибок представляют 95% доверительный интервал биномиальной пропорции).
https://doi.org/10.1371/journal.pgen.1003569.s017
(TIF)
Благодарности
Авторы благодарят Вен-Чи Сюэ за советы по статистическому анализу GWAS.
Вклад авторов
Задумал и спроектировал эксперименты: MJH IWV MTM. Проведены эксперименты: MJH IWV. Проанализированы данные: MJH IWV. Предоставленные реагенты / материалы / инструменты анализа: MJH IWV. Написал статью: MJH IWV MTM.
Ссылки
- 1.Hindorff LA, Sethupathy P, Junkins HA, Ramos EM, Mehta JP, et al. (2009) Возможные этиологические и функциональные последствия полногеномных ассоциативных локусов для болезней и признаков человека. Proc Natl Acad Sci U S A 106: 9362–9367.
- 2. Бертоне П., Столц В., Ройс Т.Э., Розовский Дж.С., Урбан А.Е. и др. (2004) Глобальная идентификация транскрибируемых последовательностей человека с помощью массивов тайлинга генома. Наука 306: 2242–2246.
- 3. Ченг Дж., Капранов П., Дренкоу Дж., Дике С., Брубакер С. и др.(2005) Транскрипционные карты 10 хромосом человека с разрешением 5 нуклеотидов. Наука 308: 1149–1154.
- 4. Капранов П., Коули С.Е., Дренков Дж., Бекиранов С., Штраусберг Р.Л. и др. (2002) Крупномасштабная транскрипционная активность в хромосомах 21 и 22. Science 296: 916–919.
- 5. Капранов П., Ченг Дж., Дике С., Никс Д.А., Дуттагупта Р. и др. (2007) Карты РНК раскрывают новые классы РНК и возможную функцию всеобъемлющей транскрипции. Science 316: 1484–1488.
- 6.Birney E, Stamatoyannopoulos JA, Dutta A, Guigo R, Gingeras TR, et al. (2007) Идентификация и анализ функциональных элементов в 1% генома человека в рамках пилотного проекта ENCODE. Природа 447: 799–816.
- 7. Mercer TR, Gerhardt DJ, Dinger ME, Crawford J, Trapnell C и др. (2012) Целевое секвенирование РНК показывает глубокую сложность человеческого транскриптома. Nat Biotechnol 30: 99–104.
- 8. Маттик Дж. С. (2009) Генетические сигнатуры некодирующих РНК.PLoS Genet 5: e1000459.
- 9. Кларк МБ, Амарал П.П., Шлезингер Ф.Дж., Дингер М.Э., Тафт Р.Дж. и др. (2011) Реальность всепроникающей транскрипции. PLoS Biol 9: e1000625 обсуждение e1001102.
- 10. Капранов П., Сен-Лоран Г. (2012) РНК темной материи: существование, функция и противоречия. Фронт Genet 3: 60.
- 11. ван Бакель Х., Нислоу С., Бленкоу Б.Дж., Хьюз Т.Р. (2011) Ответ на «Реальность всеобъемлющей транскрипции». PloS Biol 9: e1001102.
- 12. van Bakel H, Nislow C, Blencowe BJ, Hughes TR (2010) Большинство транскриптов «темной материи» связаны с известными генами. PLoS Biol 8: e1000371.
- 13. Bernstein BE, Birney E, Dunham I, Green ED, Gunter C, et al. (2012) Интегрированная энциклопедия элементов ДНК в геноме человека. Природа 489: 57–74.
- 14. Джебали С., Дэвис К.А., Меркель А., Добин А., Лассманн Т. и др. (2012) Пейзаж транскрипции в клетках человека. Природа 489: 101–108.
- 15. Wang KC, Chang HY (2011) Молекулярные механизмы длинных некодирующих РНК. Mol Cell 43: 904–914.
- 16. Кабили М.Н., Трапнелл С., Гофф Л., Козиол М., Тазон-Вега Б. и др. (2011) Интегративная аннотация больших межгенных некодирующих РНК человека раскрывает глобальные свойства и специфические подклассы. Genes Dev 25: 1915–1927.
- 17. Флокхарт Р.Дж., Вебстер Д.Е., Ку К., Маскареньяс Н., Ковальский Дж. И др. (2012) BRAFV600E ремоделирует транскриптом меланоцитов и побуждает BANCR регулировать миграцию клеток меланомы.Genome Res 22: 1006–1014.
- 18. Халил А.М., Гуттман М., Уарте М., Гарбер М., Радж А. и др. (2009) Многие большие межгенные некодирующие РНК человека связаны с модифицирующими хроматин комплексами и влияют на экспрессию генов. Proc Natl Acad Sci U S A 106: 11667–11672.
- 19. Дерриен Т., Джонсон Р., Буссотти Дж., Танзер А., Джебали С. и др. (2012) Каталог длинных некодирующих РНК человека GENCODE v7: анализ их генной структуры, эволюции и экспрессии. Genome Res 22: 1775–1789.
- 20. Харроу Дж., Фрэнкиш А., Гонсалес Дж. М., Тапанари Э., Диханс М. и др. (2012) GENCODE: справочная аннотация генома человека для проекта ENCODE. Genome Res 22: 1760–1774.
- 21. Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B (2008) Картирование и количественная оценка транскриптомов млекопитающих с помощью RNA-Seq. Нат методы 5: 621–628.
- 22. Guo H, Ingolia NT, Weissman JS, Bartel DP (2010) МикроРНК млекопитающих преимущественно действуют, снижая уровни целевых мРНК.Природа 466: 835–840.
- 23. Банфай Б., Цзя Х., Хатун Дж., Вуд Э, Риск Б и др. (2012) Длинные некодирующие РНК редко транслируются в двух линиях клеток человека. Genome Res 22: 1646–1657.
- 24. Ingolia NT, Lareau LF, Weissman JS (2011) Профилирование рибосом эмбриональных стволовых клеток мыши показывает сложность и динамику протеомов млекопитающих. Cell 147: 789–802.
- 25. Ponjavic J, Ponting CP, Lunter G (2007) Функциональность или шум транскрипции? Доказательства отбора в составе длинных некодирующих РНК.Genome Res 17: 556–565.
- 26. Ором У.А., Дерриен Т., Берингер М., Гумиредди К., Гардини А. и др. (2010) Длинные некодирующие РНК с энхансероподобной функцией в клетках человека. Ячейка 143: 46–58.
- 27. Sati S, Ghosh S, Jain V, Scaria V, Sengupta S (2012) Полногеномный анализ выявляет различные паттерны эпигенетических особенностей в длинных некодирующих локусах РНК. Nucleic Acids Res 40: 10018–10031.
- 28. Хокинс Р.Д., Хон Г.К., Ли Л.К., Нго Кью, Листер Р. и др.(2010) Четкие эпигеномные ландшафты плюрипотентных и коммитированных клонов клеток человека. Стволовая клетка клетки 6: 479–491.
- 29. Lister R, Pelizzola M, Dowen RH, Hawkins RD, Hon G, et al. (2009) Метиломы ДНК человека в базовом разрешении демонстрируют широко распространенные эпигеномные различия. Природа 462: 315–322.
- 30. Wilusz JE, Freier SM, Spector DL (2008) Процессинг 3′-конца длинной некодирующей РНК с сохранением в ядре дает тРНК-подобную цитоплазматическую РНК. Ячейка 135: 919–932.
- 31.Гутман М., Амит И., Гарбер М., Френч К., Лин М.Ф. и др. (2009) Сигнатура хроматина обнаруживает более тысячи высококонсервативных больших некодирующих РНК у млекопитающих. Природа 458: 223–227.
- 32. Ulitsky I, Shkumatava A, Jan CH, Sive H, Bartel DP (2011) Консервированная функция lincRNAs в эмбриональном развитии позвоночных, несмотря на быструю эволюцию последовательности. Cell 147: 1537–1550.
- 33. Глинский А.Б., Ма Дж., Ма С., Грант Д., Лим К.Ю. и др. (2009) Идентификация межгенных трансрегуляторных РНК, содержащих последовательность SNP, связанных с заболеванием, и нацеленных на пути прогрессирования / дифференцировки клеточного цикла при множественных распространенных заболеваниях человека.Клеточный цикл 8: 3925–3942.
- 34. Джин Дж., Сун Дж., Айзекс С.Д., Вили К.Э., Ким С.Т. и др. (2011) Человеческий полиморфизм длинных некодирующих РНК (днРНК) и связь с риском рака простаты. Канцерогенез 32: 1655–1659.
- 35. Исии Н., Одзаки К., Сато Х., Мизуно Х., Сайто С. и др. (2006) Идентификация новой некодирующей РНК, MIAT, которая создает риск инфаркта миокарда. J Hum Genet 51: 1087–1099.
- 36. Jendrzejewski J, He H, Radomska HS, Li W, Tomsic J, et al.(2012) Полиморфизм rs944289 предрасполагает к папиллярной карциноме щитовидной железы через большой межгенный некодирующий ген РНК типа опухолевого супрессора. Proc Natl Acad Sci U S A 109: 8646–8651.
- 37. Ван К.С., Ян Ю.В., Лю Б., Саньял А., Корсес-Циммерман Р. и др. (2011) Длинная некодирующая РНК поддерживает активный хроматин для координации экспрессии гомеотических генов. Природа 472: 120–124.
- 38. Dinger ME, Pang KC, Mercer TR, Mattick JS (2008) Дифференциация кодирующей белок и некодирующей РНК: проблемы и неоднозначности.PLoS Comput Biol 4: e1000176.
- 39. Окадзаки Ю., Фуруно М., Касукава Т., Адачи Дж., Боно Х. и др. (2002) Анализ транскриптома мыши на основе функциональной аннотации 60 770 полноразмерных кДНК. Природа 420: 563–573.
- 40. Lin MF, Jungreis I, Kellis M (2011) PhyloCSF: метод сравнительной геномики для различения кодирующих и некодирующих белков областей. Биоинформатика 27: i275–282.
- 41. Гуттман М., Гарбер М., Левин Дж. З., Донаги Дж., Робинсон Дж. И др.(2010) Ab initio реконструкция клеточных типоспецифичных транскриптомов у мышей выявляет консервативную мультиэкзонную структуру lincRNAs. Nat Biotechnol 28: 503–510.
- 42. Хатчинсон Дж. Н., Энсмингер А. В., Клемсон С. М., Линч С. Р., Лоуренс Дж. Б. и др. (2007) Скрининг ядерных транскриптов идентифицирует две связанные некодирующие РНК, связанные с доменами сплайсинга SC35. BMC Genomics 8: 39.
- 43. Dieci G, Fiorino G, Castelnuovo M, Teichmann M, Pagano A (2007) Транскриптом расширяющейся РНК-полимеразы III.Тенденции Genet 23: 614–622.
- 44. Тилгнер Х., Ноулз Д.Г., Джонсон Р., Дэвис К.А., Чакраборти С. и др. (2012) Глубокое секвенирование фракций субклеточной РНК показывает, что сплайсинг является преимущественно котранскрипционным в геноме человека, но неэффективен для днРНК. Genome Res 22: 1616–1625.
- 45. Koehler R, Issac H, Cloonan N, Grimmond SM (2011) Уникальный: ресурс сопоставимости для секвенирования коротких тегов. Биоинформатика 27: 272–274.
- 46. Ян Л., Дафф М.О., Грейвли Б.Р., Кармайкл Г.Г., Чен Л.Л. (2011) Полногеномная характеристика неполиаденилированных РНК.Геном Биол 12: R16.
лекций25
лекций259 марта 2005
Лекция 25
Чтение: Глава 8
VI. Гены
A. Синтез белка (Центральный Догма)
B. Геномы
C. Регуляция генов
Деятельность, п.е. транскрипция гены строго регулируются. В нашем обсуждении мы поместим гены в три категории, основанные на их регуляции: конститутивные гены, индуцибельные гены и гены с молчанием. Учредительные гены — это те которые всегда активны. Примером являются гены рибосом. Они есть постоянно транскрибируется, потому что рибосомы постоянно нужны для синтеза белка. Индуцибельные гены — это гены, у которых есть переменные активность, в зависимости от потребностей клетки.Например, белки-переносчики глюкозы, которые мышечные клетки производят в ответ на инсулин — продукт индуцибельных генов. Инсулин стимулирует их деятельность. Заглушенные гены — это те гены, которые были навсегда обращены выключенный. Например, некоторые гены, необходимые для функционирования нервных клеток, будут заглушается в мышечных клетках. Подавление гена — часть процесса клетки приобретают различную идентичность.
1. Промоторы и транскрипция Постановление
Гены состоят из разных частей.В последовательность оснований, которая кодирует аминокислоты в белке, — это всего лишь одно из этих. Другая часть всех генов — это промотор . Это сегмент ДНК, расположенный выше области, которая транскрибируется в информационная РНК. Это часть гена, с которой связывается РНК-полимераза. и начинает копирование последовательности гена в информационную РНК.
Транскрипция генов контролируется белками, которые связываются с промотором или рядом с ним. Некоторые белки будут способствуют связыванию РНК-полимеразы и тем самым транскрипции ген.Другие белки будут препятствовать связыванию РНК-полимеразы и таким образом предотвратить транскрипцию гена. Эти белки называются «генные регуляторные белки». (Вы увидите, что они называются транскрипцией факторов и регуляторных белков в вашем тексте, и есть много других названия для них, в зависимости от того, что делает белок. Мы назовем их все «генные регуляторные белки»). Кодируются регуляторные белки генов для «регуляторных генов». Регуляторные белки генов часто связываются с более одного гена, что означает, что регуляторный ген может одновременно регулируют активность наборов генов.
Некоторые регуляторные белки генов связываются с места, отличные от промотора гена, но все же влияют на его транскрипция. Например, некоторые белки связываются с «энхансерами», которые — это участки ДНК, расположенные на сотни оснований выше промоутер. Считается, что эти области могут закручиваться так, что они взаимодействуют с промотором и улучшают связывание РНК-полимеразы для транскрипции.
2.Интроны, экзоны и альтернативы сращивание
В области гена, который транскрибируется в информационную РНК, есть сегменты ДНК, которые не код для аминокислотной последовательности. Эти сегменты называются «интронами», что является сокращением от «промежуточных последовательностей». Части гена последовательность, которая транслируется в аминокислотную последовательность, называется «экзоны». Экзоны и интроны копируются РНК в информационную РНК. полимеразы, но интроны вырезаются путем редактирования ферментов до информационная РНК покидает ядро.Последовательности интронов вырезаны из молекула матричной РНК и экзоны сплайсированы вместе.
Иногда экзоны гена соединены несколькими разными способами, что приводит к разным виды белков. Этот процесс называется «альтернативным сращиванием». Альтернативный сплайсинг — это средство, с помощью которого гены могут регулироваться после транскрипция происходит. Это также способ для клеток производить больше белки, чем у них есть гены. Поскольку синтез белка происходил была выдвинута гипотеза о том, что один ген кодирует один белок.В целом это так, но альтернативное сращивание — одно из возможных решений. исключение из этого правила.
Что такое транскрипция? Улучшение доступности, SEO и UX
17 августа 2018 г. BY JACLYN LEDUC
Обновлено: 16 марта 2021 г.
взрослых американцев проводят 11 часов в день, подключаясь к средствам массовой информации, и почти 6 из них тратятся на просмотр видео. Возможно, вы оказались в ситуации, когда вам не терпится посмотреть видео или послушать аудио, но сейчас не время и не место для этого.К счастью, есть еще один способ потреблять желаемый контент: транскрипция.
Что такое транскрипция?
Транскрипция — это процесс преобразования речи или звука в письменный документ. Скрытые титры привязаны к видео по времени, а расшифровка — это просто текст без информации о времени. Транскрипция — отличный вариант сделать программы, содержащие только аудио, такие как подкасты и радиошоу, более доступными для глухих и слабослышащих людей. Когда дело доходит до видео, транскрипция является отличным дополнением к субтитрам; тем не менее, это не считается заменой на основании законов и стандартов доступности.
Дословная и чистая стенограмма чтения
Существует две основные практики транскрипции: дословная и чистая. Дословная практика транскрибирует текст дословно и включает в себя всю речь и высказывания, которые произносит говорящий. Слова-заполнители, такие как «ммм», речевые ошибки и сленговые слова, включаются в расшифровку стенограммы. Это обычно полезно для сценариев, где все создается намеренно, и это более чем вероятно имеет отношение к сюжету или сюжетной линии.
Чистое чтение — это текстовый формат, исключающий речевые ошибки, слова-вставки и любые другие непреднамеренные высказывания говорящего. Эта практика полезна для интервью, публичных выступлений и других средств массовой информации без сценария.
При любой транскрипции следует придерживаться некоторых рекомендаций. Убедившись, что каждая стенограмма совпадает со звуком, и что в ней указаны разные выступающие, будет иметь большое значение для вашей аудитории. Когда дело доходит до транскрипции, ключевыми факторами являются точность и ясность!
Теперь, когда мы ответили на вопрос «Что такое транскрипция?», Давайте углубимся в дополнительную информацию, которая поможет вам на пути к пониманию транскрипции в целом.
Форматы стенограммы
Существует несколько форматов файлов с расшифровками, каждый из которых имеет свои преимущества и преимущества.
HTML
Если вы хотите, чтобы ваша стенограмма размещалась на веб-странице, этот формат файла для вас. Вы можете сделать свой HTML-текст совместимым с программами чтения с экрана. Программы чтения с экрана преобразуют цифровой текст в синтезированную речь и полезны для слепых или слабовидящих людей, а также людей с ограниченными когнитивными функциями или способностями к обучению. Этот формат позволяет программе чтения с экрана преобразовывать цифровой текст в синтезированную речь.
Word (.doc) или текстовый (.txt) документ
В то время как текстовые и текстовые документы предоставляют текстовую версию вашей расшифровки, документ .txt вообще не имеет форматирования. В отличие от текстового документа файл документа Word представляет собой отформатированную версию текста, и ее можно редактировать или сохранять в неизменном виде. Наряду с простым документом Word вы также можете создавать документы с отметками времени и документы с отметками SMPTE.
PDF (.pdf)
Как и документ Word, PDF-файл представляет собой простой текстовый файл с форматированием.Основное отличие состоит в том, что конечный пользователь не имеет возможности редактировать файл. PDF-файлы также легко загрузить в Интернет.
Как расшифровать видео
Кто угодно может транскрибировать видео. Исходя из ваших потребностей, вы можете решить расшифровать свой контент самостоятельно или обратиться в профессиональную службу транскрипции.
Внутренняя расшифровка
Вы не поверите, но расшифровать видео и аудио самостоятельно или самостоятельно довольно просто. Все, что вам нужно сделать, это прослушать видео или аудио и вручную ввести озвученный контент в текстовый редактор.Это недорогой способ обеспечить расшифровку всех ваших медиафайлов, но, как вы понимаете, это трудоемкий процесс.
К счастью, есть определенные инструменты, которые немного облегчат это бремя. Программное обеспечение автоматического распознавания речи (ASR), такое как Dragon или Camtasia, запустит процесс за вас, хотя будет много ошибок. Идея состоит в том, что для редактирования неточной расшифровки требуется гораздо меньше труда, чем для того, чтобы начать с самого начала самостоятельно. Вы даже можете пойти дальше и использовать программное обеспечение для транскрипции, такое как Express Scribe, которое предназначено для повышения эффективности процесса транскрипции.
Служба профессиональной транскрипции
Для тех, кто создает или владеет большими медиа-библиотеками, и у кого нет пропускной способности для создания стенограмм внутри компании, профессиональная служба транскрипции может быть подходящей для вас. Ваши медиафайлы будут возвращены вам во всех форматах вывода, избавляя от необходимости вручную конвертировать файлы. Хотя эта услуга стоит дорого, она сэкономит вам время и силы. Вы можете сесть и расслабиться или сосредоточиться на других вещах, пока ваше видео и аудио записываются издалека.
Достаточно ли транскрипции для удовлетворения требований законодательства?
Хотя транскрипция — отличный способ сделать видео более доступным для любой аудитории, сама по себе этого недостаточно для соблюдения требований законодательства.
Закон об американцах с ограниченными возможностями (ADA)
Закон об американцах с ограниченными возможностями (ADA) — это широкий закон о борьбе с дискриминацией в отношении лиц с ограниченными возможностями. Это требует, чтобы в отношении общения эквивалентный опыт был доступен для глухих или слабослышащих зрителей.Простая расшифровка текста не позволяет зрителю синхронно следить за текстом и изображениями, что потенциально может сбить с толку зрителя и не дать ему понять контекст должным образом. По этой причине для видеоконтента необходимы скрытые субтитры.
Закон о реабилитации
Раздел 508 Закона о реабилитации требует соблюдения стандартов WCAG 2.0 AA. В связи с этим, чтобы все предварительно записанное и живое видео соответствовало закону, должны быть включены скрытые субтитры.Согласно WCAG 2.0 и обновленному 2.1, стандарты уровня A требуют расшифровки стенограммы для всего аудиоконтента.
Законы о доступности делают использование скрытых субтитров обязательным, а стенограмма сама по себе не соответствует требованиям закона. В конечном итоге цель состоит в том, чтобы обеспечить равный доступ к контенту для всех человек.
Преимущества транскрипции
Транскрипция дает три основных преимущества: доступность, удобство для пользователя (UX) и поисковая оптимизация (SEO).
Доступность
Для глухих и слабослышащих людей расшифровки стенограммы недостаточно для создания эквивалентного впечатления от просмотра. Однако это может помочь более четко передать устное сообщение. Для слепых и слабовидящих людей они могут использовать стенограммы, чтобы описывать визуальные аспекты медиа. И скрытые субтитры, и стенограммы работают вместе, чтобы создать доступный для всех медиафайл. Замечательно то, что вы можете использовать видео- и аудиозаписи для создания скрытых субтитров.
Пользовательский опыт (UX)
Стенограммыулучшают взаимодействие с пользователем, предоставляя посетителям альтернативный способ взаимодействия с вашим видео- или аудиоконтентом.СМИ можно сделать более привлекательными, включив интерактивную расшифровку стенограммы и поиск по списку воспроизведения. Если опыт будет лучше, то пользователи будут проводить больше времени на вашей странице или сайте, что положительно повлияет на SEO страницы.
Согласно национальному исследованию Университета штата Орегон, стенограммы влияют на опыт студентов в высшем образовании. Более 99% студентов посещают занятия, в которых видео включено в курсовую работу, что делает видео частью их учебного процесса.Данные показывают, что 81% участников используют стенограммы в качестве учебного пособия, чтобы помочь сохранить информацию, найти информацию и в качестве учебного пособия.
Поисковая оптимизация (SEO)
Поисковые системы не могут сканировать или индексировать видео или аудио контент. Имея расшифровку стенограммы, это позволяет поисковым системам делать именно это. В целом, это сделает ваш видео- или аудиоконтент более доступным для поиска и более легким для вашей аудитории. Это также поможет вашей странице ранжироваться по более разнообразным ключевым словам, которые могут быть включены в вашу расшифровку.
Интерактивные стенограммы
Интерактивные стенограммы, также известные как синхронизированные по времени стенограммы, представляют собой еще один способ взаимодействия пользователей с вашим контентом. Каждое слово стенограммы ведет себя как отдельная ссылка, которая при нажатии ведет прямо к тому месту в видео, когда оно произносится. Существует также возможность поиска в расшифровке стенограммы, что упрощает поиск определенных ключевых слов и тем.
Поиск по плейлисту
Поиск по плейлисту — это функция, которая объединяет видео в плейлист вместе с их транскриптами.Панель поиска позволяет пользователям выполнять поиск по ключевым словам во всей видеотеке. Включение этой функции на ваш сайт имеет множество преимуществ, таких как лучшее взаимодействие с пользователем, повышенная доступность, а также более высокая эффективность и организация мультимедиа.
Исследование SEO: эта американская жизнь
This American Life (TAL) — общественное радио-шоу и один из самых популярных подкастов. TAL транслирует более 500 станций и имеет почти 2,1 миллиона пользователей. Шоу уделяет существенное внимание своему веб-сайту, предоставляя своим слушателям бесплатную потоковую передачу всех шоу.
TAL намеревалась расшифровать 100% своей аудиотеки, чтобы увеличить входящий трафик и ссылки, улучшить взаимодействие с пользователем и сделать их контент более доступным.
После расшифровки всего архива TAL были достигнуты впечатляющие результаты. Входящие ссылки увеличились на 3,89%, уникальные посетители увеличились на 4,18%, а обычный поиск увеличился на 6,68%.
This American Life — истинное свидетельство того, как транскрипция может положительно повлиять на ваши цели и рост.
Теперь, когда вы знаете о преимуществах транскрипции, вы можете начать делать свой видео- и аудиоконтент более удобным и доступным для поиска.
3Play Media может помочь вам с вашими потребностями в транскрипции!2.1: Обзор транскрипции — биология LibreTexts
Что такое транскрипция?
Учтите, что все клетки в многоклеточном организме возникли в результате деления из одной оплодотворенной яйцеклетки и, следовательно, все имеют одинаковую ДНК.При делении этой исходной оплодотворенной яйцеклетки в случае человека образуется более триллиона клеток к тому времени, когда из этой яйцеклетки рождается ребенок (это большая репликация ДНК!). Тем не менее, мы также знаем, что ребенок — это не гигантский шар из триллиона идентичных клеток, но он имеет множество различных типов клеток, из которых состоят ткани, такие как кожа, мышцы, кости и нервы. Как клетки с идентичной ДНК оказались такими разными?
Ответ кроется в экспрессии генов, то есть в процессе использования информации ДНК.Хотя все клетки ребенка имеют одинаковую ДНК, каждый разный тип клеток использует разные подмножества генов в этой ДНК, чтобы направлять синтез отличительного набора РНК и белков. Первым шагом в экспрессии генов является транскрипция, процесс копирования информации из последовательностей ДНК в последовательности РНК. Этот процесс также известен как ДНК-зависимый синтез РНК. Когда последовательность ДНК транскрибируется, только одна из двух цепей ДНК копируется в РНК, когда эта РНК кодирует белок, он известен как информационная РНК (мРНК).
Рисунок \ (\ PageIndex {1} \): структура одной цепи РНКВажные особенности транскрипции
- Вся РНК, мРНК, а также тРНК, рРНК, микроРНК и другие производятся путем транскрипции.
- Только одна цепь ДНК используется в качестве матрицы ферментами, называемыми РНК-полимеразами
- РНК синтезируется от 5 ‘до 3’.
- РНК-полимеразам не нужны праймеры для начала транскрипции.
- Четыре рибонуклеотидтрифосфата (rNTP) — это АТФ, GTP, UTP и CTP.
- РНК-полимеразы начинают транскрипцию с последовательностей ДНК, называемых промоторами.
- РНК-полимеразы заканчивают транскрипцию на последовательностях, называемых терминаторами.
При транскрипции РНК-полимераза использует только одну цепь ДНК, называемую цепочкой-матрицей, гена, чтобы катализировать синтез комплементарной антипараллельной цепи РНК. РНК-полимеразы используют предшественники рибозонуклеотидтрифосфата (NTP), в отличие от ДНК-полимераз, которые используют предшественники нуклеотидов дезоксирибозы (dNTP) (сравнение на странице 1.1: Структура ДНК). Кроме того, РНК включают нуклеотиды урацила (U) в цепи РНК вместо нуклеотидов тимина (T), используемых в ДНК. РНК-полимеразы отличаются от ДНК-полимераз тем, что не требуют праймеров. С помощью факторов инициации транскрипции РНК-полимераза определяет местонахождение сайта начала транскрипции гена и начинает синтез новой цепи РНК с нуля, соединяя два рибонуклеотида, которые комплементарны первым двум основаниям цепи матрицы.
Обзор этапов транскрипции
Основными этапами транскрипции являются инициация, удлинение и завершение.Здесь мы можем идентифицировать несколько последовательностей ДНК, характеризующих ген. Промотор — это сайт связывания РНК-полимеразы. Обычно он расположен в 5 ’или выше сайта начала транскрипции. Связывание РНК-полимеразы размещает фермент рядом с сайтом начала транскрипции, где он начинает раскручивать двойную спираль и начинает синтез новой РНК. Транскрибируемая серая область ДНК на каждой из трех панелей представляет собой единица транскрипции гена. Сайты терминации обычно расположены на 3 ’или ниже транскрибируемой области гена.Условно, выше относится к ДНК 5 ’к заданной контрольной точке на ДНК (например, сайту начала транскрипции гена). Далее относится к ДНК 3 ’к заданной контрольной точке на ДНК.
Рисунок \ (\ PageIndex {2} \): три этапа транскрипции. (Авторское право)РНК-полимераза
Построение цепи РНК очень похоже на построение цепи ДНК. Это неудивительно, зная, что ДНК и РНК — очень похожие молекулы. Какой фермент осуществляет транскрипцию? Транскрипция катализируется ферментом РНК-полимеразой.«РНК-полимераза» — это общий термин для фермента, который производит РНК. Есть много различных РНК-полимераз.
Рисунок \ (\ PageIndex {3} \): РНК-полимераза T7 (синий), создающая РНК (зеленый) с использованием ДНК-матрицы (коричневый)Как и ДНК-полимеразы, РНК-полимеразы синтезируют новые цепи только в направлении от 5 ‘к 3′, но потому что они производят РНК, они используют рибонуклеотиды (т.е. нуклеотиды РНК), а не дезоксирибонуклеотиды. Рибонуклеотиды соединяются точно так же, как дезоксирибонуклеотиды, то есть 3’OH последнего нуклеотида в растущей цепи присоединяется к 5’-фосфату на входящем нуклеотиде.
Одно важное различие между ДНК-полимеразами и РНК-полимеразами состоит в том, что последним не требуется праймер для начала образования РНК. Как только РНК-полимеразы оказываются в нужном месте, чтобы начать копирование ДНК, они просто начинают создавать РНК, связывая вместе нуклеотиды РНК, комплементарные матрице ДНК.
Это, конечно, подводит нас к очевидному вопросу — откуда РНК-полимеразы «знают», где начать копирование ДНК. В отличие от ситуации с репликацией, когда каждый нуклеотид родительской ДНК должен в конечном итоге быть скопирован, транскрипция, как мы уже отметили, копирует только выбранные гены в РНК в любой момент времени.Что указывает РНК-полимеразе, где начать копирование ДНК для создания транскрипта? Сигналы в ДНК указывают РНК-полимеразе, где она должна начать (и закончить) транскрипцию. Эти сигналы представляют собой особые последовательности в ДНК, которые распознаются РНК-полимеразой или белками, которые помогают РНК-полимеразе определять, где она должна связывать ДНК, чтобы начать транскрипцию. Последовательность ДНК, с которой связывается РНК-полимераза, чтобы начать транскрипцию, называется промотором.
Промотор обычно расположен выше гена, который он контролирует.Это означает, что в цепи ДНК, на которой расположен ген, промоторная последовательность находится «перед» геном. Помните, что по соглашению последовательности ДНК читаются от 5 ‘до 3’. Таким образом, промотор находится в 5 ‘от начальной точки транскрипции.
Также обратите внимание, что промотор, как говорят, «контролирует» ген, с которым он связан. Это связано с тем, что экспрессия гена зависит от связывания РНК-полимеразы с промоторной последовательностью для начала транскрипции. Если РНК-полимераза и ее вспомогательные белки не связываются с промотором, ген не может быть транскрибирован и, следовательно, не будет экспрессироваться.
Рисунок \ (\ PageIndex {4} \): Промоторные последовательностиЧто особенного в промоторной последовательности? Пытаясь ответить на этот вопрос, ученые изучили множество генов и окружающих их последовательностей. Имеет смысл, что, поскольку одна и та же РНК-полимераза должна связываться с множеством разных промоторов, промоторы должны иметь некоторое сходство в своих последовательностях. Разумеется, общие паттерны последовательностей присутствовали во многих промоторах. Сначала мы рассмотрим прокариотические промоторы. При исследовании прокариотических генов обычно выявлялись следующие особенности:
- Сайт начала транскрипции (это основание в ДНК, через которое спарен первый нуклеотид РНК). Последовательность
- A -10: это область длиной 6 п.н. с центром примерно в 10 п.н. выше стартового сайта. Консенсусная последовательность в этом положении — TATAAT. Другими словами, если вы отсчитываете от сайта начала транскрипции, который по соглашению называется +1, последовательность, обнаруженная в -10 в большинстве изученных промоторов, будет TATAAT). Последовательность
- A -35: это последовательность примерно на 35 пар оснований выше начала транскрипции. Консенсусная последовательность в этом положении — TTGACA.
Какое значение имеют эти последовательности? Оказывается, что последовательности -10 и -35 распознаются и связываются субъединицей прокариотической РНК-полимеразы до того, как может начаться транскрипция.
РНК-полимераза E. coli, например, имеет субъединицу, называемую сигма (σ) субъединицей (или сигма-фактором), в дополнение к основной полимеразе, которая является частью фермента, который фактически производит РНК. Вместе сигма-субъединица и основная полимераза составляют то, что называется холоферментом РНК-полимеразы .Сигма-субъединица полимеразы может распознавать и связываться с последовательностями -10 и -35 в промоторе, таким образом располагая РНК-полимеразу в нужном месте для инициации транскрипции. Как только начинается транскрипция, основная полимераза и сигма-субъединица разделяются, при этом основная полимераза продолжает синтез РНК, а сигма-субъединица перемещается, чтобы сопроводить другую базовую молекулу полимеразы к промотору. Сигма-субъединицу можно рассматривать как своего рода проводника, который приводит полимеразу к ее «месту» на промоторе.
Рисунок \ (\ PageIndex {5} \): Инициирование транскрипции в E. coliКак уже упоминалось, цепь РНК, комплементарная матрице ДНК, создается РНК-полимеразой путем присоединения 5′-фосфата входящего рибонуклеотида. к 3’OH на последнем нуклеотиде растущей цепи РНК. Как полимераза знает, где остановиться? Последовательность нуклеотидов, называемая терминатором, является сигналом для РНК-полимеразы, чтобы остановить транскрипцию и отделиться от матрицы.
Хотя процесс синтеза РНК у эукариот такой же, как и у прокариот, у эукариот есть некоторые дополнительные проблемы, о которых следует помнить.Во-первых, у эукариот матрица ДНК существует в виде хроматина, где ДНК тесно связана с гистонами и другими белками. Следовательно, «упаковка» ДНК должна быть открыта, чтобы позволить РНК-полимеразе получить доступ к матрице в области, которая должна быть транскрибирована.
Второе отличие состоит в том, что эукариоты имеют несколько РНК-полимераз, а не одну, как в бактериальных клетках. Различные полимеразы транскрибируют разные гены. Например, РНК-полимераза I транскрибирует гены рибосомной РНК, а РНК-полимераза III копирует гены тРНК.РНК-полимераза, на которой мы сосредоточимся больше всего, — это РНК-полимераза II, которая транскрибирует гены, кодирующие белок, для образования мРНК.
Рисунок \ (\ PageIndex {6} \): Сборка базального транскрипционного комплекса и инициация транскрипцииВсем трем эукариотическим РНК-полимеразам нужны дополнительные белки, чтобы помочь им начать транскрипцию. У прокариот РНК-полимераза сама по себе может инициировать транскрипцию (помните, что сигма-субъединица является субъединицей прокариотической РНК-полимеразы). Дополнительные белки, необходимые для эукариотических РНК-полимераз, называются факторами транскрипции.
Наконец, в эукариотических клетках транскрипция отделена во времени и пространстве от трансляции. Транскрипция происходит в ядре, и продуцируемые мРНК обрабатываются дальше, прежде чем они будут отправлены в цитоплазму. Синтез (трансляция) белка происходит в цитоплазме. В прокариотических клетках мРНК могут транслироваться по мере того, как они выходят из матрицы ДНК, и поскольку ядра нет, транскрипция и синтез белка происходят в одном клеточном компартменте.
Подобно генам прокариот, гены эукариот также имеют промоторы.Эукариотические промоторы обычно имеют ТАТА-бокс, последовательность примерно из 25 пар оснований перед началом транскрипции, которая распознается и связывается белками, которые помогают РНК-полимеразе правильно позиционировать себя для начала транскрипции. (У некоторых эукариотических промоторов нет ТАТА-боксов, и вместо этого есть другие последовательности распознавания, которые помогают РНК-полимеразе найти место на ДНК, где она находится на ДНК, где она связывается и инициирует транскрипцию.)
Мы отметили ранее, что эукариотические РНК-полимеразы нуждаются в дополнительных белках для связывания промоторов и начала транскрипции.Какие дополнительные белки необходимы для начала транскрипции? Общие факторы транскрипции — это белки, которые помогают эукариотическим РНК-полимеразам находить сайты начала транскрипции и инициировать синтез РНК. Мы сосредоточимся на факторах транскрипции, которые помогают РНК-полимеразе II. Эти факторы транскрипции называются TFIIA, TFIIB и так далее (TF = фактор транскрипции, II = РНК-полимераза II, а буквы обозначают отдельные факторы транскрипции).
Транскрипция у эукариот требует, чтобы общие факторы транскрипции и РНК-полимераза образовали комплекс в ТАТА-боксе, называемый базальным комплексом транскрипции или комплексом инициации транскрипции.Это минимальное требование для транскрибирования любого гена. Первым шагом в образовании этого комплекса является связывание ТАТА-бокса с помощью фактора транскрипции, называемого ТАТА-связывающим белком или ТВР. Связывание ТВР заставляет ДНК изгибаться в этом месте и принимать структуру, подходящую для связывания дополнительных факторов транскрипции и РНК-полимеразы. Как показано на рисунке слева, ряд различных общих факторов транскрипции вместе с РНК-полимеразой (Pol II) образуют комплекс в ТАТА-боксе.
Заключительным этапом сборки базального транскрипционного комплекса является связывание общего фактора транскрипции, называемого TFIIH. TFIIH — это многофункциональный белок, который обладает геликазной активностью (т.е. он способен открывать двойную спираль ДНК), а также киназной активностью.


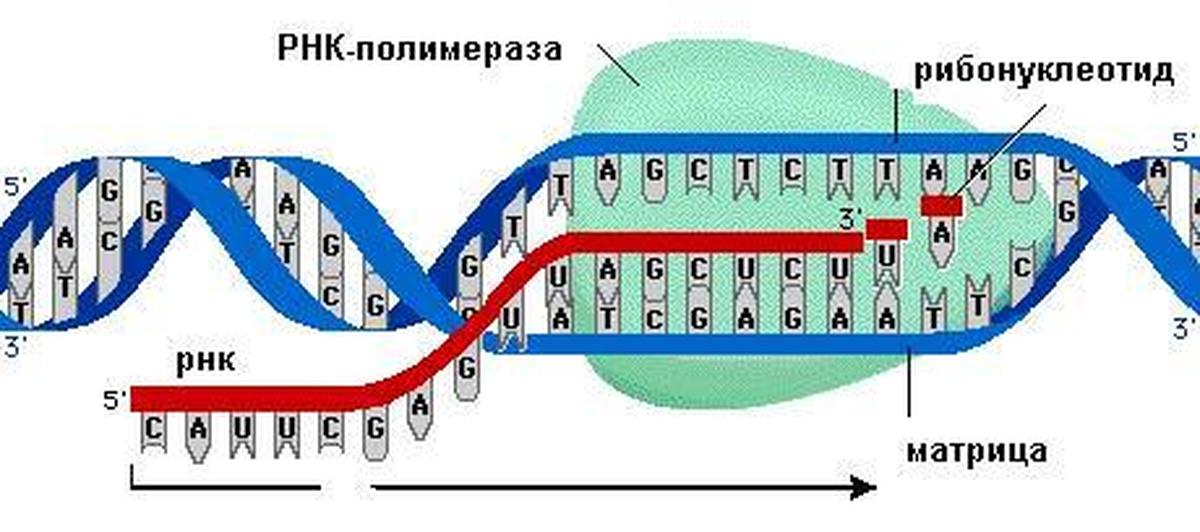

 А. Брокгауза и И.А. Ефрона
А. Брокгауза и И.А. Ефрона
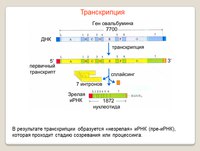

 У меня были мои родители, о которых я рассказывал немного ранее. Я не слишком много рассказывал о своем деде, но он оказал на меня большое влияние. Мне сильно повезло, поскольку моя мама была очень молода, каждое лето, начиная лет с четырех, я проводил у дедушки с бабушкой, чтобы мама немного отдохнула от меня – целое лето».
У меня были мои родители, о которых я рассказывал немного ранее. Я не слишком много рассказывал о своем деде, но он оказал на меня большое влияние. Мне сильно повезло, поскольку моя мама была очень молода, каждое лето, начиная лет с четырех, я проводил у дедушки с бабушкой, чтобы мама немного отдохнула от меня – целое лето». Ммм…(сглатывает) и потом, кроме того, у меня была куча ролевых моделей, которые я перепробовал в жизни. Да, гм, (сглатывает) некоторые из моих учителей в школе River Oaks Elementary, о которых вы упомянули. У меня были, гм, у меня были мои родители, о которых я рассказывал немного ранее. Я не слишком много рассказывал о своем деде, но он оказал на меня большое влияние. Мне сильно повезло (сглатывает), поскольку моя мама была очень молода, каждое лето, даже… начиная лет с четырех, я проводил у дедушки с бабушкой, просто чтобы мама немного отдохнула от меня – целое лето».
Ммм…(сглатывает) и потом, кроме того, у меня была куча ролевых моделей, которые я перепробовал в жизни. Да, гм, (сглатывает) некоторые из моих учителей в школе River Oaks Elementary, о которых вы упомянули. У меня были, гм, у меня были мои родители, о которых я рассказывал немного ранее. Я не слишком много рассказывал о своем деде, но он оказал на меня большое влияние. Мне сильно повезло (сглатывает), поскольку моя мама была очень молода, каждое лето, даже… начиная лет с четырех, я проводил у дедушки с бабушкой, просто чтобы мама немного отдохнула от меня – целое лето».